Yaohai Bio-Pharma bereitet circRNA auf Basis des PIE-Systems (Anordnung von Exonen und Intronen) vor, das sich auf die selbstspaltende Funktion von Typ-I-Intronen stützt, um die RNA-Zyklisierung zu erreichen. Die PIE-Struktur wird mit dem T4 td-Gen oder dem fischen tRNA-Vorgängergen entworfen, und die Anordnung ist wie folgt:
Das RNA-Intron und das unterstützende Exonfragment werden in zwei Teile unterteilt (5' End und 3' End), wobei die 5'-Endsequenz an das Ende der Zielsequenz übertragen wird, die 3'-Endsequenz vor die Zielsequenz eingefügt wird und die Zielgensequenz in der Mitte eingefügt wird.
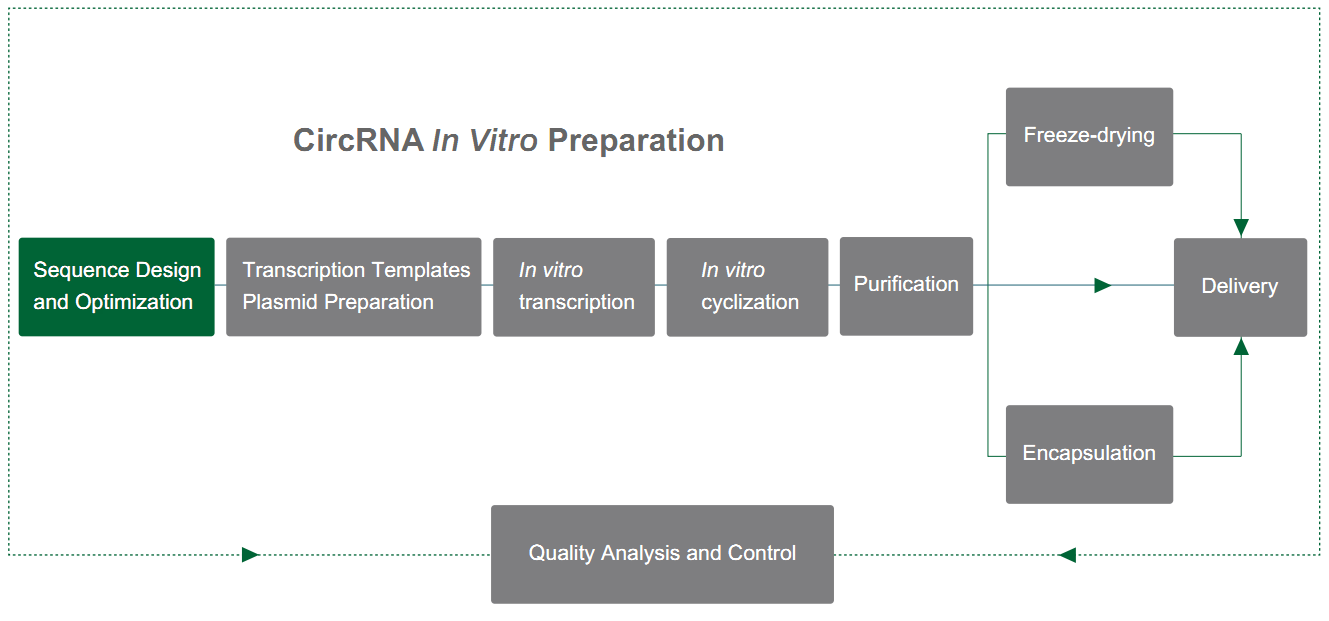
Unter der Katalyse von GTP führt die PIE-Struktur zur Zyklierung von Sequenzen außerhalb von Introns. In Kombination mit einer vernünftigen Strategie zur Steigerung der Zyklierungsrate kann Yaohai Bio-Pharma eine Zyklierung von Sequenzen bis zu 4 kb mit einer Zyklierungsrate von über 80 % erreichen.
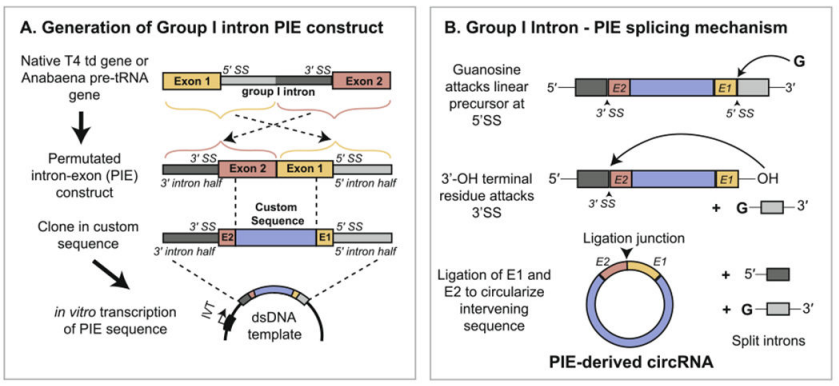
Abbildung 1. In-vitro-Zyklierung von circRNA basierend auf dem PIE-System
Dienstleistungen Details
| Prozess |
Optionale Dienstleistungen |
Service Details |
Lieferzeit (Arbeitstag) |
| design und Optimierung von circRNA-Sequenzen |
Design und Optimierung von Codesequenzen |
CDS-Sequenzalignierung
CDS-Codonoptimierung
|
1 |
| Entwurf und Optimierung von nicht kodierenden Sequenzen |
Design und Optimierung von Intron- und Exon-Sequenzen
Design und Optimierung von homologen Armsequenzen
Design und Optimierung von Spacer-Sequenzen
|
1-2 |
Häufige Strategien für die Design von mRNA-Sequenzen
| CircRNA-Komponenten |
Biologische Funktionen |
Optimierungsstrategien |
| Zwei-terminal intron- und exon-Sequenzen |
GTP-katalysierte intron Selbstsplicing zur Zyklierung von Sequenzen außerhalb des Introns. |
Entworfen auf der Basis des T4 td Gens oder des FischöltRNA-Präcursor-Gens. |
| Code |
IRES |
Interner Ribosomenanerkennungsplatz, der die Übersetzung von circRNA reguliert. |
Screening von internen Ribosomen-Eintritts-Sites (IRES)-Sequenzen aus verschiedenen viralen Quellen, z. B. EMCV, CVB3-Quellen. |
| CDS |
Protein-codierende Bereiche, Sequenzen, die für Antigene, Antikörper oder andere funktionelle Proteine codieren. |
Codon-Optimierung erhöht das Übersetzungslevel; bestimmte nicht optimale Codons könnten eine Rolle im Protein-Falten spielen. |
| Nicht-codierend |
Nicht-codierende Sequenzen |
Zielgerichtete miRNAs oder Proteine zur Ausübung der Gen- oder Proteinkontrolle. |
Zielgerichtete spezifische Bindungsstellen für miRNAs oder Proteine können die Sequenzen der Bindungsstelle wiederholen. |
Unsere Funktionen
- Optimierte PIE-Cyclisierungs-Systemkombination mit vernünftigen Optimierungsstrategien, um eine Cyclisierungsrate von über 80 % zu erreichen;
- Kooperation des innovativen CDS-Optimierungsteams mit einem professionellen KI-Algorithmus-Team zur Vervollständigung der Codon-Optimierung des CDS-Bereichs;
- Reifes und perfektes circRNA kann mit hoher Zyklierungseffizienz, hoher Stabilität und hoher Translations-effizienz erreicht werden.
Fallstudie
Yaohai Bio-Pharma hat das Kontrollprodukt Enhanced Green Fluorescent Protein (eGFP) circRNA lanciert, welches auf Basis des PIE-Systems die Zyklierung von RNA realisiert.
Mit einem konventionellen Transfektionsreagenz wird das eGFP circRNA in 293T-Zellen transfektiert, und nach 24 Stunden kann ein eGFP (grün) Fluoreszenzsignal detektiert werden, welches nach 48 Stunden verstärkt wird. Das Fluoreszenzsignal kann auch noch am 7. und 14. Tag nach der Transfektion detektiert werden.
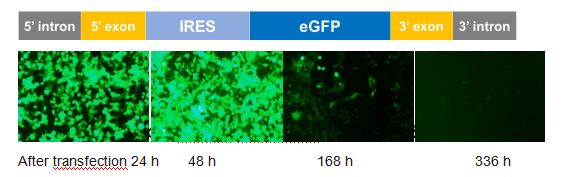
In-vitro-Ausdruckvalidierung von eGFP circRNA

 EN
EN
 AR
AR
 HR
HR
 CS
CS
 DA
DA
 NL
NL
 FI
FI
 FR
FR
 DE
DE
 EL
EL
 IT
IT
 JA
JA
 KO
KO
 NO
NO
 PL
PL
 PT
PT
 RO
RO
 RU
RU
 ES
ES
 SV
SV
 IW
IW
 ID
ID
 LV
LV
 LT
LT
 SR
SR
 SK
SK
 SL
SL
 UK
UK
 VI
VI
 ET
ET
 HU
HU
 TH
TH
 TR
TR
 FA
FA
 AF
AF
 MS
MS
 BE
BE
 MK
MK
 UR
UR
 BN
BN