ヤオハイ・バイオファーマは、PIEシステム(エクソンとイントロンのアラインメント)に基づいてcircRNAを準備します。これは、タイプIイントロンの自己スプライシング機能に依存してRNAのサイクリゼーションを達成します。PIE構造はT4 td遺伝子または魚類tRNA前駆体遺伝子を使用して設計され、その配置は次の通りです:
RNAイントロンとサポートエクソン断片は2つの部分(5'末端と3'末端)に分けられ、5'末端配列はターゲット配列の末尾に転送され、3'末端配列はターゲット配列の前に挿入され、ターゲット遺伝子配列は中央に挿入されます。
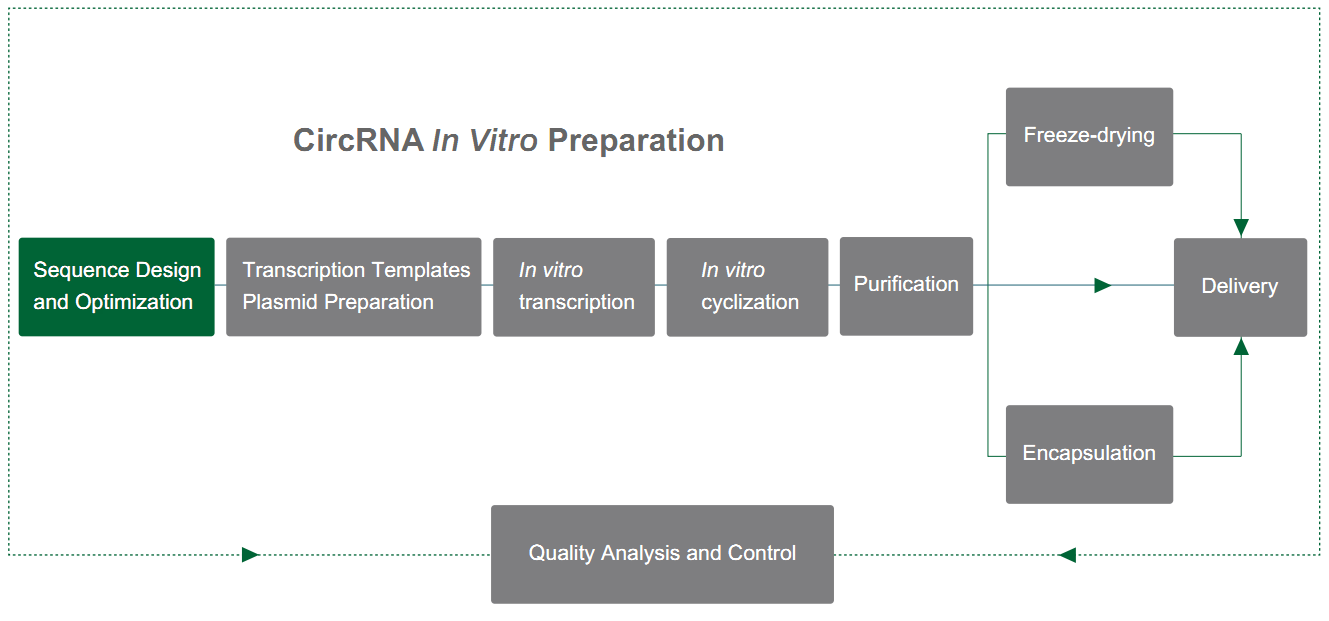
GTPの触媒作用の下で、PIE構造はイントロン以外の配列のサイクリゼーションを導きます。合理的なサイクリゼーション効率向上戦略と組み合わせることで、ヤオハイ・バイオファーマは80%以上のサイクリゼーション効率で最大4 kbの配列のサイクリゼーションを実現できます。
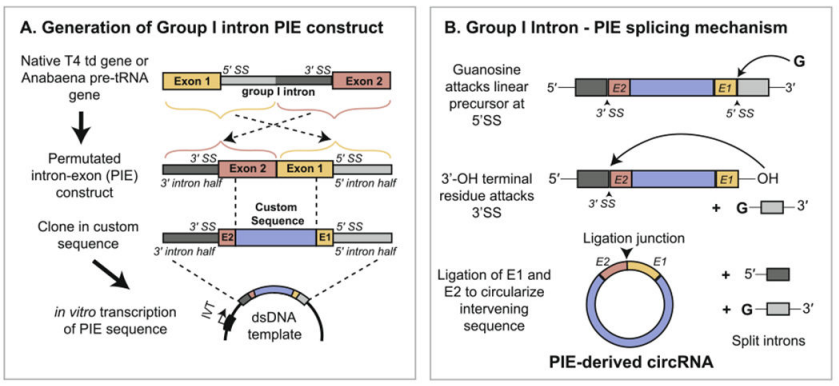
図1. PIEシステムに基づくcircRNAの体外循環
| プロセス | オプションサービス | サービス詳細 | 納期(営業日) |
| circRNA配列設計および最適化 | コーディング配列の設計と最適化 |
CDS配列のアラインメント CDSコドンの最適化 |
1 |
| ノンコーディング配列の設計と最適化 |
イントロンおよびエクソン配列の設計と最適化 ホモロガスアーム配列の設計と最適化 Spacer配列の設計と最適化 |
1-2 |
| CircRNAの構成要素 | 生物学的機能 | 最適化戦略 | |
| 二端イントロンおよびエクソン配列 | GTP触媒によるイントロンの自己スプライシングで、イントロン外の配列をサイクリングする。 | T4 td遺伝子または魚油tRNA前駆体遺伝子に基づいて設計されたもの。 | |
| コード化 | IRES | CircRNAの翻訳を制御するリボソーム内部認識サイト。 | 異なるウイルス由来の内部リボソーム進入サイト(IRES)配列のスクリーニング、例えばEMCV、CVB3由来。 |
| CDS | タンパク質をコードする領域、抗原、抗体またはその他の機能的なタンパク質をコードする配列。 | コドン最適化は翻訳のレベルを向上させる;非最適なコドンの一部はタンパク質の折り畳みに役割を果たす可能性がある。 | |
| ノンコーディング | ノンコーディング配列 | 遺伝子やタンパク質の調節を行うためにmiRNAやタンパク質をターゲットにする。 | MiRNAやタンパク質の特定の結合部位をターゲットにして、結合部位の配列を繰り返すことができる。 |
Yaohai Bio-Pharmaは、PIEシステムに基づいてRNAのサイクリゼーションを実現する制御製品である強化緑色蛍光タンパク質(eGFP)circRNAを発売しました。
従来のトランスフェクション試薬を使用して、eGFP circRNAを293T細胞にトランスフェクトすると、24時間後にeGFP(緑色)の蛍光信号を検出でき、48時間後にはさらに強化されます。トランスフェクション後の7日目と14日目にも依然として蛍光信号を検出できます。
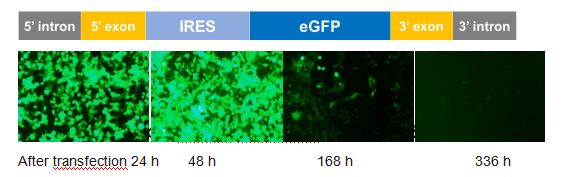
EGFP circRNAの体外発現確認