Yaohai Bio-Pharma przygotowuje circRNA w oparciu o system PIE (urównanie eksonów i intronów), który opiera się na funkcji samosplicingu intronów typu I w celu osiągnięcia cyklizacji RNA. Strukturę PIE zaprojektowano przy użyciu genu T4 td lub genu prekursora rybiego tRNA, a układ jest następujący:
Intron RNA i fragment egzonu pomocniczego są podzielone na dwie części (koniec 5' i koniec 3'), gdzie sekwencja końca 5' jest przenoszona na ogon sekwencji docelowej, sekwencja końca 3' jest wstawiana z przodu sekwencję docelową, a docelowa sekwencja genu jest wstawiana w środku.
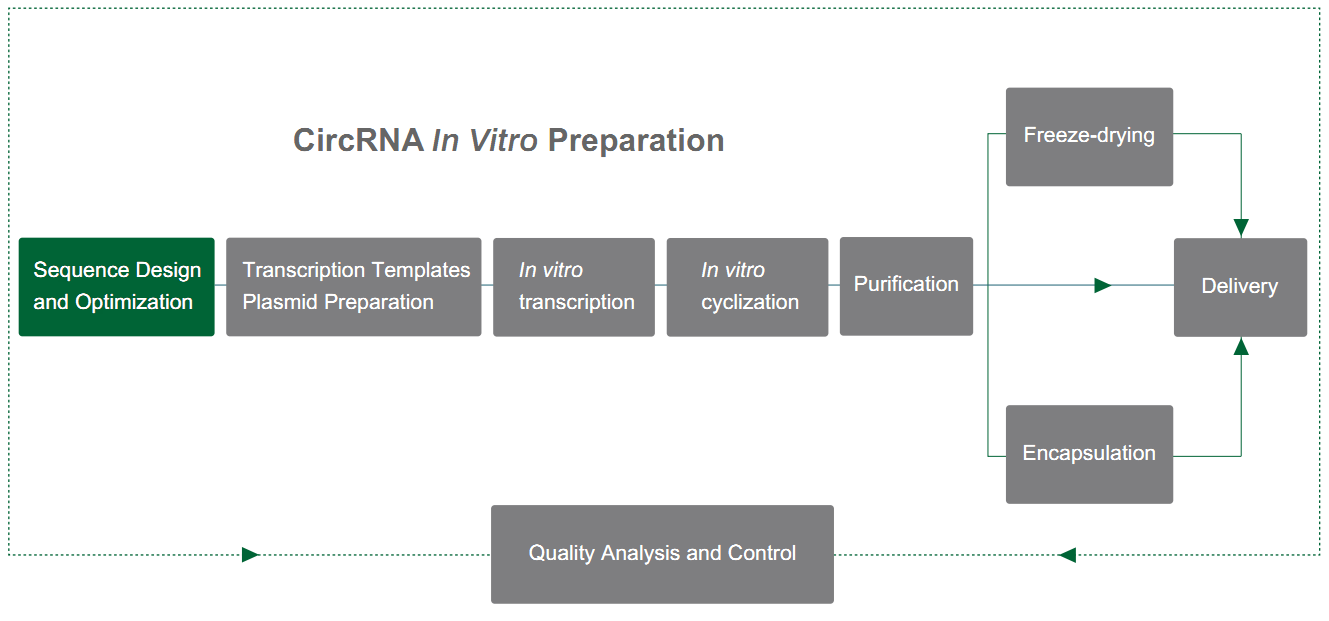
Pod wpływem katalizy GTP struktura PIE prowadzi do cyklizacji sekwencji innych niż introny. W połączeniu z rozsądną strategią zwiększania szybkości cyklizacji, Yaohai Bio-Pharma może osiągnąć cyklizację sekwencji o wielkości do 4 kb przy szybkości cyklizacji przekraczającej 80%.
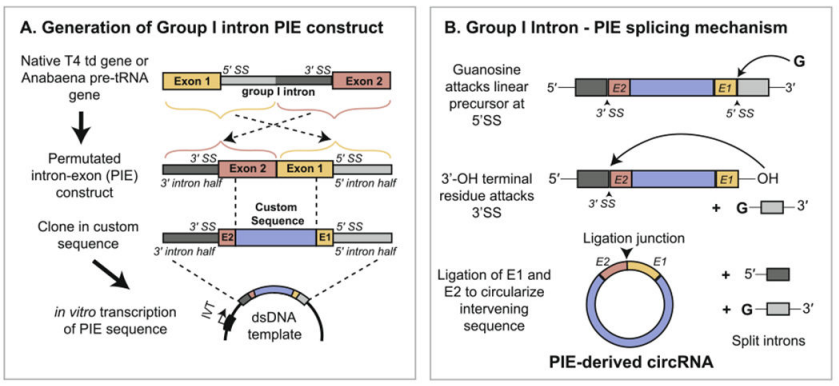
Rycina 1. Cyrkulacja in vitro circRNA w oparciu o system PIE
Szczegóły usług
| Przetwarzanie |
Usługa opcjonalna |
Szczegóły usługi |
Termin dostawy (dzień roboczy) |
| Projektowanie i optymalizacja sekwencji circRNA |
Projektowanie i optymalizacja sekwencji kodujących |
Dopasowanie sekwencji CDS
Optymalizacja kodonów CDS
|
1 |
| Projektowanie i optymalizacja sekwencji niekodujących |
Projektowanie i optymalizacja sekwencji intronów i eksonów
Projektowanie i optymalizacja sekwencji ramion homologicznych
Projektowanie i optymalizacja sekwencji odstępników
|
1-2 |
Typowe strategie projektowania sekwencji mRNA
| Składniki CircRNA |
Funkcje biologiczne |
Strategie optymalizacji |
| Dwukońcowe sekwencje intronów i eksonów |
Samosplatanie intronów katalizowane GTP w celu cyklizacji sekwencji poza intronem. |
Zaprojektowane zgodnie z genem T4 td lub genem prekursora tRNA oleju rybnego. |
| Kodowanie |
IRES |
Wewnętrzne miejsce rozpoznawania rybosomów, które reguluje translację circRNA. |
Skrining sekwencji wewnętrznego miejsca wejścia rybosomu (IRES) z różnych źródeł wirusowych, np. źródeł EMCV, CVB3. |
| CDS |
Regiony kodujące białka, sekwencje kodujące antygeny, przeciwciała lub inne białka funkcjonalne. |
Optymalizacja kodonów zwiększa poziom translacji; pewne nieoptymalne kodony mogą odgrywać rolę w fałdowaniu białek. |
| Bez kodowania |
Sekwencje niekodujące |
Celuj w miRNA lub białka, aby wywierać regulację genów lub białek. |
Celowanie w określone miejsca wiązania miRNA lub białek może powtarzać sekwencje miejsca wiązania. |
Nasze funkcje
- Zoptymalizowany system cyklizacji PIE Połączenie z rozsądnymi strategiami optymalizacji w celu osiągnięcia współczynnika cyklizacji powyżej 80%;
- Najnowocześniejszy zespół ds. optymalizacji CDS Współpraca z profesjonalnym zespołem algorytmów AI w celu zakończenia optymalizacji kodonów regionu CDS;
- Dojrzały i doskonały proces circRNA można osiągnąć dzięki wysokiej wydajności cyklizacji, wysokiej stabilności i wysokiej wydajności translacji.
Case study
Firma Yaohai Bio-Pharma wprowadziła na rynek produkt kontrolny wzmocniony białkiem zielonej fluorescencji (eGFP) circRNA, oparty na systemie PIE w celu osiągnięcia cyklizacji RNA.
Przy użyciu konwencjonalnego odczynnika do transfekcji, eGFP circRNA transfekuje się komórki 293T, a sygnał fluorescencyjny eGFP (zielony) można wykryć po 24 godzinach, który zostanie wzmocniony po 48 godzinach. Sygnał fluorescencyjny można nadal wykryć 7. i 14. dnia po transfekcja.
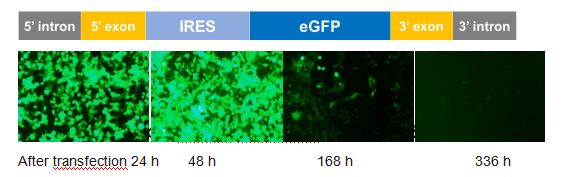
Walidacja ekspresji in vitro circRNA eGFP

 EN
EN
 AR
AR
 HR
HR
 CS
CS
 DA
DA
 NL
NL
 FI
FI
 FR
FR
 DE
DE
 EL
EL
 IT
IT
 JA
JA
 KO
KO
 NIE
NIE
 PL
PL
 PT
PT
 RO
RO
 RU
RU
 ES
ES
 SV
SV
 IW
IW
 ID
ID
 LV
LV
 LT
LT
 SR
SR
 SK
SK
 SL
SL
 UK
UK
 VI
VI
 ET
ET
 HU
HU
 TH
TH
 TR
TR
 FA
FA
 AF
AF
 MS
MS
 BE
BE
 MK
MK
 UR
UR
 BN
BN