Yaohai Bio-Pharma przygotowuje circRNA na podstawie systemu PIE (wyrównanie eksonów i intronów), który opiera się na funkcji samo-splotowej intronów typu I w celu osiągnięcia cykliczności RNA. Struktura PIE jest projektowana za pomocą genu T4 td lub prekursora tRNA ryb, a układ jest następujący:
Intron RNA i wspierający fragment eksona są dzielone na dwie części (końcówka 5' i końcówka 3'), gdzie sekwencja końcowej 5' jest przenoszona na koniec sekwencji docelowej, sekwencja końcowej 3' jest wstawiana przed sekwencję docelową, a sekwencja genu docelowego jest wstawiana w środku.
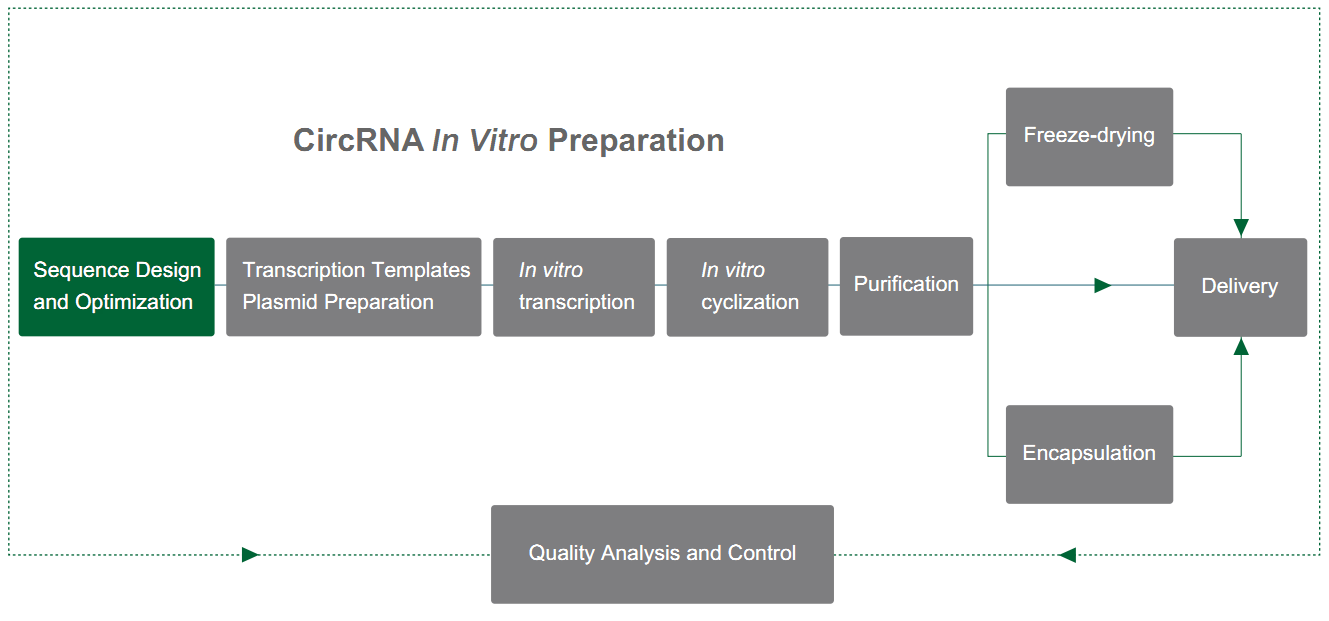
W wyniku katalizy GTP struktura PIE prowadzi do cyklowania sekwencji innych niż introny. W połączeniu z rozsądną strategią wzmacniania tempa cyklowania, Yaohai Bio-Pharma może osiągnąć cyklowanie sekwencji o długości do 4 kb przy tempie cyklowania przekraczającym 80%.
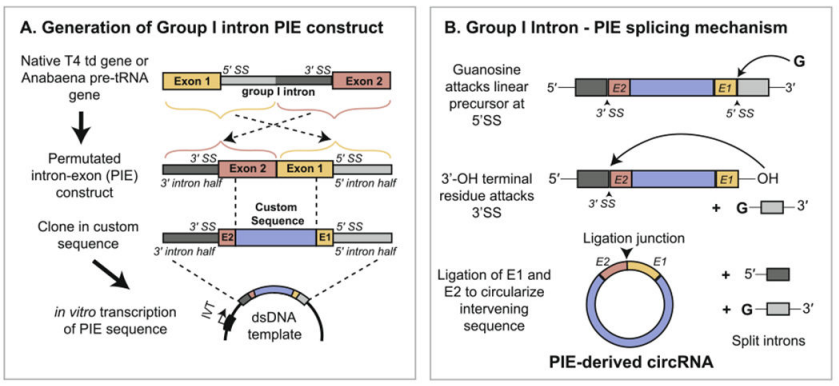
Rysunek 1. In vitro cyrkulacja circRNA oparta na systemie PIE
Szczegóły usług
| Proces |
Opcjonalne Usługi |
Szczegóły usług |
Okres dostawy (dzień roboczy) |
| projektowanie i optymalizacja sekwencji circRNA |
Projektowanie i optymalizacja sekwencji kodujących |
Wyrównanie sekwencji CDS
Optymalizacja kodonów sekwencji CDS
|
1 |
| Projektowanie i optymalizacja sekwencji niekodujących |
Projektowanie i optymalizacja sekwencji intronów i ektronów
Projektowanie i optymalizacja sekwencji ramy homologicznej
Projektowanie i optymalizacja sekwencji spacerowych
|
1-2 |
Powszechne Strategie Projektowania Sekwencji mRNA
| Komponenty circRNA |
Funkcje Biologiczne |
Strategie Optymalizacji |
| Sekwencje intronów i ekstronów dwusterownikowych |
Autokatalizowana samozakręcanina intronów poprzez GTP do cyklicznego łączenia sekwencji poza intronem. |
Zaprojektowano na podstawie genu T4 td lub prekursora tRNA z oleju rybnego. |
| Kodujące |
IRES |
Wewnętrzny punkt rozpoznawania rybosomu, który reguluje tłumaczenie circRNA. |
Ekranowanie sekwencji wewnętrznego wejścia rybosomowego (IRES) z różnych wirusowych źródeł, np. EMCV, CVB3. |
| CDS |
Regiony kodujące białka, sekwencje kodujące antygeny, przeciwciała lub inne funkcjonalne białka. |
Optymalizacja kodonów zwiększa poziom tłumaczenia; niektóre nieoptymalne kodony mogą odgrywać rolę w fałdowaniu białek. |
| Niekodujący |
Sekwencje niekodujące |
Celowanie w miRNY lub białka w celu wywierania regulacji genu lub białka. |
Celowanie w określone miejsca wiązania dla miRNY lub białek może powtarzać sekwencje miejsca wiązania. |
Nasze cechy
- Zoptymalizowany System Cyklizacji PIE za pomocą rozsądnego podejścia do optymalizacji, aby osiągnąć stopień cyklizacji większy niż 80%;
- Nowoczesna współpraca zespołu optymalizacji CDS z profesjonalnym zespołem algorytmów sztucznej inteligencji w celu ukończenia optymalizacji kodonów regionu CDS;
- Dojrzały i doskonały proces circRNA można osiągnąć za pomocą wysokiej efektywności cyklicznej, wysokiej stabilności i wysokiej efektywności translacji.
Badanie przypadków
Yaohai Bio-Pharma wprowadziła produkt kontrolny zintensyfikowany białkowy fluorescencyjny (eGFP) circRNA, który opiera się na systemie PIE do realizacji cykliczności RNA.
Korzystając z konwencjonalnego środka transfekcyjnego, eGFP circRNA jest transfekowany do komórek 293T, a sygnał fluorescencyjny eGFP (zielony) może być wykryty po 24 godzinach, a po 48 godzinach wzmacnia się. Sygnał fluorescencyjny można nadal wykrywać w 7. i 14. dniu po transfekcji.
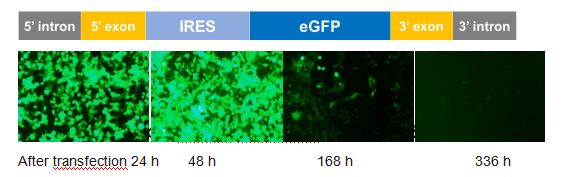
Weryfikacja wyrażania się in vitro eGFP circRNA

 EN
EN
 AR
AR
 HR
HR
 CS
CS
 DA
DA
 NL
NL
 FI
FI
 FR
FR
 DE
DE
 EL
EL
 IT
IT
 JA
JA
 KO
KO
 NO
NO
 PL
PL
 PT
PT
 RO
RO
 RU
RU
 ES
ES
 SV
SV
 IW
IW
 ID
ID
 LV
LV
 LT
LT
 SR
SR
 SK
SK
 SL
SL
 UK
UK
 VI
VI
 ET
ET
 HU
HU
 TH
TH
 TR
TR
 FA
FA
 AF
AF
 MS
MS
 BE
BE
 MK
MK
 UR
UR
 BN
BN