Yaohai Bio-Pharma prépare le circRNA basé sur le système PIE (alignement des exons et des introns), qui s'appuie sur la fonction d'auto-épissage des introns de type I pour réaliser la cyclisation de l'ARN. La structure PIE est conçue à l’aide du gène T4 td ou du gène précurseur de l’ARNt fishy, et la disposition est la suivante :
L'intron d'ARN et le fragment d'exon de support sont divisés en deux parties (terminale 5' et terminale 3'), où la séquence terminale 5' est transférée à la queue de la séquence cible, la séquence terminale 3' est insérée à l'avant de la séquence cible. séquence cible, et la séquence du gène cible est insérée au milieu.
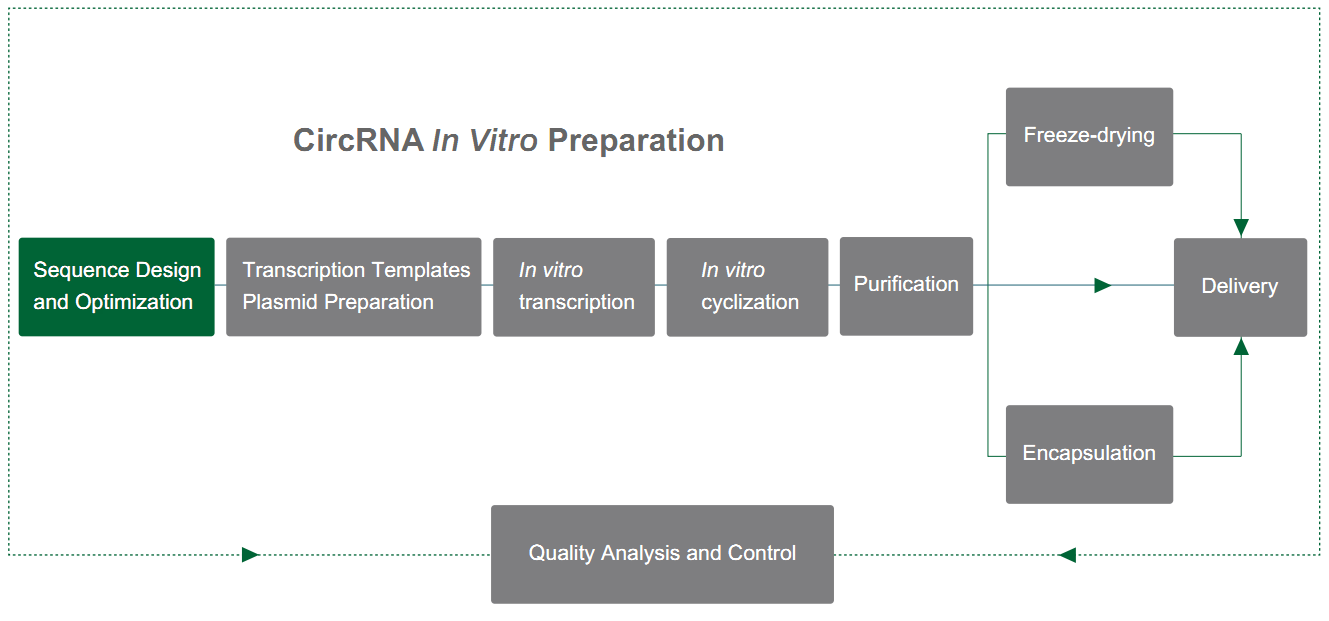
Sous la catalyse du GTP, la structure PIE conduit à la cyclisation de séquences autres que les introns. Combiné à une stratégie raisonnable d'amélioration du taux de cyclisation, Yaohai Bio-Pharma peut réaliser la cyclisation de séquences jusqu'à 4 kb avec un taux de cyclisation de plus de 80 %.
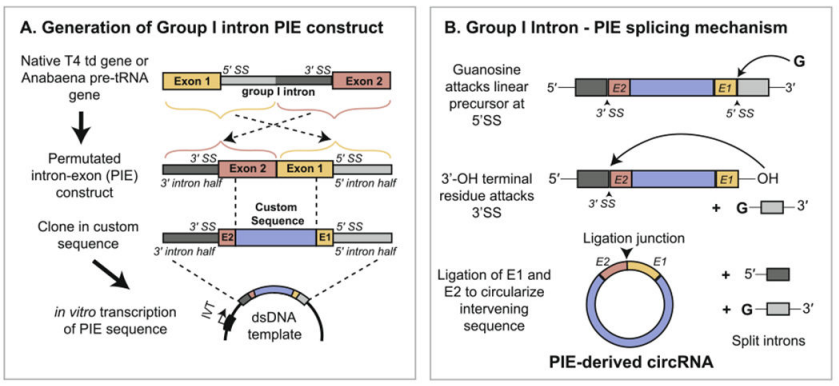
Figure 1. Circulation in vitro de circARN basée sur le système PIE
Détails des services
| Processus |
Service en option |
Détails du service |
Délai de livraison (jour ouvrable) |
| conception et optimisation de séquences circRNA |
Conception et optimisation de séquences de codage |
Alignement de séquence CDS
Optimisation des codons CDS
|
1 |
| Conception et optimisation de séquences non codantes |
Conception et optimisation de séquences d'introns et d'exons
Conception et optimisation de séquences de bras homologues
Conception et optimisation de séquences d’espacement
|
1-2 |
Stratégies courantes pour la conception de séquences d'ARNm
| Composants CircRNA |
Fonctions biologiques |
Stratégies d'optimisation |
| Séquences d'intron et d'exon à deux bornes |
Auto-épissage d'intron catalysé par GTP pour la cyclisation de séquences en dehors de l'intron. |
Conçu selon le gène T4 td ou le gène précurseur de l'ARNt de l'huile de poisson. |
| Codage |
IRE |
Site interne de reconnaissance des ribosomes qui régule la traduction des circARN. |
Criblage des séquences du site d'entrée interne du ribosome (IRES) provenant de différentes sources virales, par exemple sources EMCV, CVB3. |
| CDS |
Régions codant pour des protéines, séquences codant pour des antigènes, des anticorps ou d'autres protéines fonctionnelles. |
L'optimisation des codons augmente le niveau de traduction ; certains codons non optimaux peuvent jouer un rôle dans le repliement des protéines. |
| Non-codage |
Séquences non codantes |
Ciblez les miARN ou les protéines pour exercer une régulation génique ou protéique. |
Le ciblage de sites de liaison spécifiques pour les miARN ou les protéines peut répéter les séquences du site de liaison. |
Nos offres
- Combinaison optimisée du système de cyclisation PIE avec des stratégies d'optimisation raisonnables pour atteindre un taux de cyclisation de plus de 80 % ;
- Équipe d'optimisation CDS de pointe Coopération avec l'équipe professionnelle d'algorithmes d'IA pour achever l'optimisation des codons de la région CDS ;
- Le circRNA à processus mature et parfait peut être obtenu avec une efficacité de cyclisation élevée, une stabilité élevée et une efficacité de traduction élevée.
Étude de cas
Yaohai Bio-Pharma a lancé le circRNA de la protéine fluorescente verte améliorée (eGFP), produit de contrôle, qui est basé sur le système PIE pour réaliser la cyclisation de l'ARN.
À l'aide d'un réactif de transfection conventionnel, le circARN eGFP est transfecté dans des cellules 293T et le signal fluorescent eGFP (vert) peut être détecté après 24 heures, qui sera amélioré après 48 heures. Le signal fluorescent peut toujours être détecté le 7ème jour et le 14ème jour après transfection.
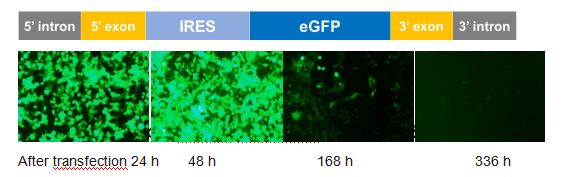
Validation de l'expression in vitro du circARN eGFP

 EN
EN
 AR
AR
 HR
HR
 CS
CS
 DA
DA
 NL
NL
 FI
FI
 FR
FR
 DE
DE
 EL
EL
 IT
IT
 JA
JA
 KO
KO
 NON
NON
 PL
PL
 PT
PT
 RO
RO
 RU
RU
 ES
ES
 SV
SV
 IW
IW
 ID
ID
 LV
LV
 LT
LT
 SR
SR
 SK
SK
 SL
SL
 UK
UK
 VI
VI
 ET
ET
 HU
HU
 TH
TH
 TR
TR
 FA
FA
 AF
AF
 MS
MS
 BE
BE
 MK
MK
 UR
UR
 BN
BN