Yaohai Bio-Pharma prepara circRNA basandosi sul sistema PIE (allineamento di esoni ed introni), che si avvale della funzione di autocatalisi degli introni di tipo I per ottenere la ciclizzazione dell'RNA. La struttura PIE è progettata utilizzando il gene T4 td o il gene precursore del tRNA ittico, e l'ordinamento è il seguente:
L'introne RNA e il frammento esonico di supporto sono divisi in due parti (estremità 5' e 3'), dove la sequenza 5' terminale viene trasferita alla coda della sequenza obiettivo, la sequenza 3' terminale viene inserita prima della sequenza obiettivo, e la sequenza del gene obiettivo viene inserita al centro.
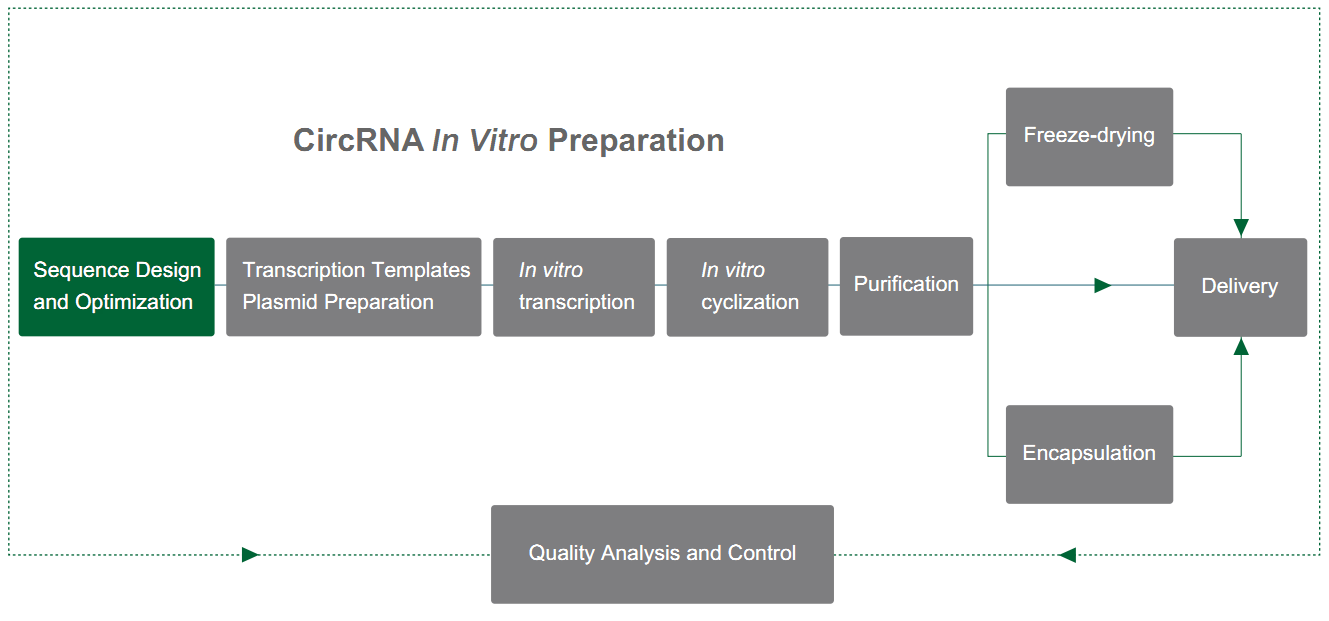
Sotto la catalisi del GTP, la struttura PIE conduce alla ciclizzazione delle sequenze diverse dagli introni. Combinata con una strategia ragionevole di miglioramento del tasso di ciclizzazione, Yaohai Bio-Pharma può ottenere la ciclizzazione di sequenze fino a 4 kb con un tasso di ciclizzazione superiore al 80%.
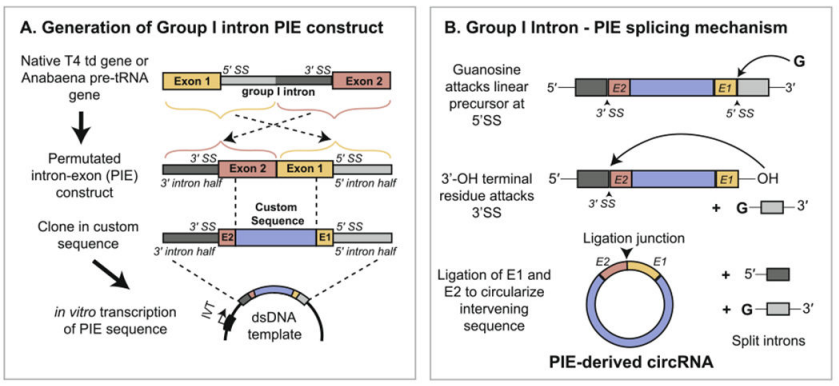
Figura 1. Circolazione in vitro di circRNA basata sul sistema PIE
Dettagli dei Servizi
| Processo |
Servizio Opzionale |
Dettagli del servizio |
Periodo di consegna (giornata lavorativa) |
| progettazione e ottimizzazione della sequenza di circRNA |
Progettazione e ottimizzazione delle sequenze codificanti |
Allineamento della sequenza CDS
Ottimizzazione del codone CDS
|
1 |
| Progettazione e ottimizzazione di sequenze non codificanti |
Progettazione e ottimizzazione delle sequenze degli introni ed esoni
Progettazione e ottimizzazione delle sequenze degli bracci omologhi
Progettazione e ottimizzazione della sequenza dello spaziatore
|
1-2 |
Strategie comuni per la progettazione della sequenza di mRNA
| Componenti del CircRNA |
Funzioni biologiche |
Strategie di ottimizzazione |
| Sequenze di introni e esoni terminali |
Auto-incisione catalizzata da GTP dell'introne per la ciclizzazione delle sequenze esterne all'introne. |
Progettato in base al gene T4 td o al gene precursore del tRNA dell'olio di pesce. |
| Codifica |
IRES |
Sito di riconoscimento interno del ribosoma che regola la traduzione del circRNA. |
Screening delle sequenze del sito di ingresso interna del ribosoma (IRES) da fonti virali diverse, ad esempio EMCV, fonti CVB3. |
| CDS |
Regioni codificanti proteine, sequenze che codificano anticorpi, antigeni o altre proteine funzionali. |
L'ottimizzazione dei codoni aumenta il livello di traduzione; alcuni codoni non ottimali possono svolgere un ruolo nella piegatura delle proteine. |
| Non codificante |
Sequenze non codificanti |
Indirizzano miRNA o proteine per esercitare una regolazione genica o proteica. |
Il targeting di siti di legame specifici per miRNA o proteine può ripetere le sequenze del sito di legame. |
Le nostre caratteristiche
- Sistema di Ciclizzazione PIE Ottimizzato combinato con strategie di ottimizzazione razionali per raggiungere un tasso di ciclizzazione superiore al 80%;
- Collaborazione con un Team di Ottimizzazione CDS all'avanguardia e un team di algoritmi AI professionale per completare l'ottimizzazione dei codoni della regione CDS;
- Un processo maturo e perfetto per circRNA può essere realizzato con alta efficienza di ciclizzazione, alta stabilità ed elevata efficienza di traduzione.
Studio di caso
Yaohai Bio-Pharma ha lanciato il prodotto di controllo proteina fluorescente verde migliorata (eGFP) circRNA, che si basa sul sistema PIE per realizzare la ciclizzazione dell'RNA.
Utilizzando un reagente di trasfusione convenzionale, l'eGFP circRNA viene trasfuso in cellule 293T e dopo 24 ore può essere rilevato il segnale fluorescente di eGFP (verde), il quale si intensificherà dopo 48 ore. Il segnale fluorescente può ancora essere rilevato al settimo giorno e al quattordicesimo giorno dopo la trasfusione.
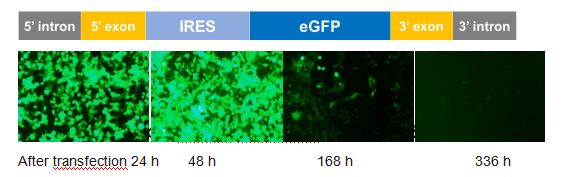
Validazione dell'espressione in vitro di eGFP circRNA

 EN
EN
 AR
AR
 HR
HR
 CS
CS
 DA
DA
 NL
NL
 FI
FI
 FR
FR
 DE
DE
 EL
EL
 IT
IT
 JA
JA
 KO
KO
 NO
NO
 PL
PL
 PT
PT
 RO
RO
 RU
RU
 ES
ES
 SV
SV
 IW
IW
 ID
ID
 LV
LV
 LT
LT
 SR
SR
 SK
SK
 SL
SL
 UK
UK
 VI
VI
 ET
ET
 HU
HU
 TH
TH
 TR
TR
 FA
FA
 AF
AF
 MS
MS
 BE
BE
 MK
MK
 UR
UR
 BN
BN