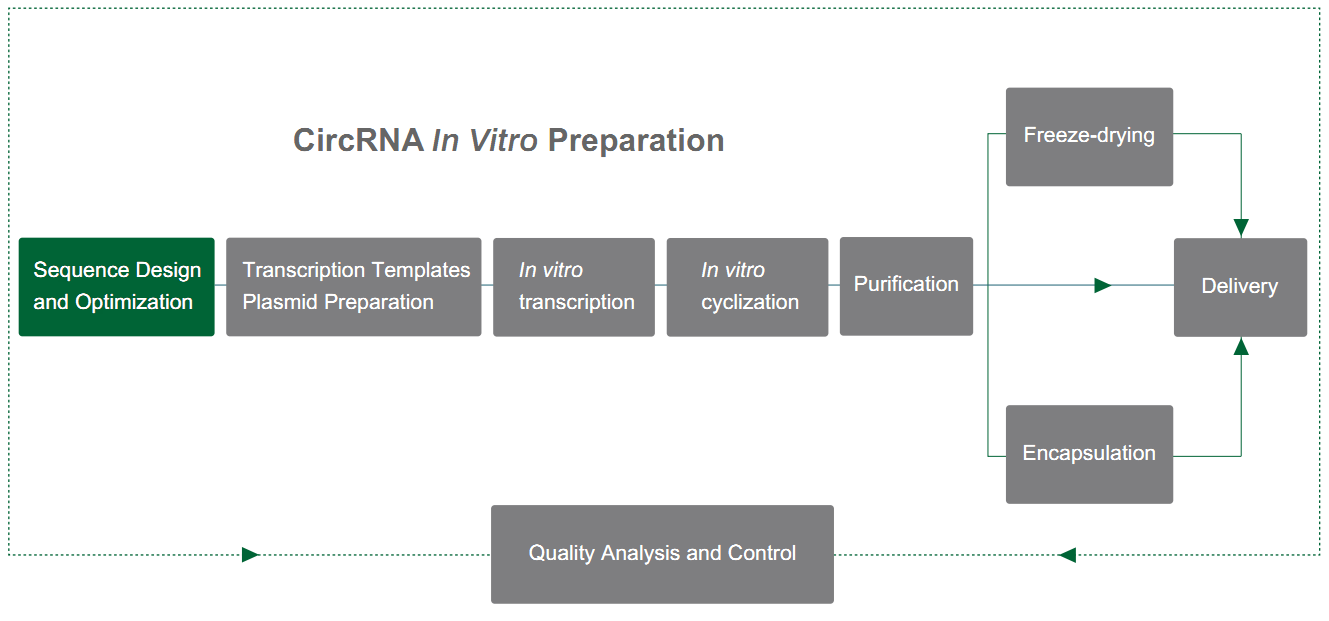
Yaohai Bio-Pharma prepara circRNA basado en el sistema PIE (alineación de exones e intrones), que se basa en la función de autoempalme de los intrones de tipo I para lograr la ciclación del ARN. La estructura PIE se diseña utilizando el gen T4 td o el gen precursor del ARNt de pescado, y la disposición es la siguiente:
El intrón de ARN y el fragmento del exón de soporte se dividen en dos partes (terminal 5' y terminal 3'), donde la secuencia terminal 5' se transfiere a la cola de la secuencia objetivo, la secuencia terminal 3' se inserta en la parte frontal de la secuencia objetivo, y la secuencia del gen objetivo se inserta en el medio.
Bajo la catálisis de GTP, la estructura PIE conduce a la ciclación de secuencias distintas de los intrones. Combinado con una estrategia de mejora razonable de la tasa de ciclación, Yaohai Bio-Pharma puede lograr la ciclación de secuencias de hasta 4 kb con una tasa de ciclación de más del 80%.
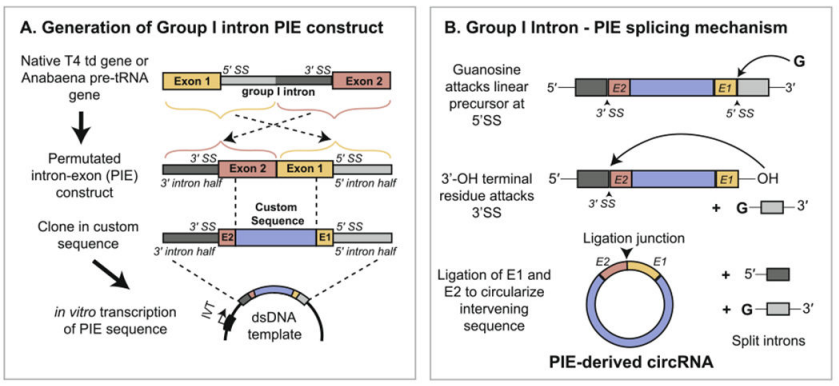
Figura 1. Circulación in vitro de circRNA basada en el sistema PIE
Detalles de servicios
| Proceso | Servicio opcional | Detalle de Servicios | Plazo de entrega (día laborable) |
| Diseño y optimización de secuencias de circRNA. | Diseño y optimización de secuencias de codificación. | Alineación de secuencia CDS Optimización de codones CDS | 1 |
| Diseño y optimización de secuencias no codificantes. | Diseño y optimización de secuencias de intrones y exones. Diseño y optimización de secuencias de brazos homólogos. Diseño y optimización de secuencia espaciadora. | 1 - 2 |
Estrategias comunes para el diseño de secuencias de ARNm
| Componentes de ARN circular | Funciones biológicas | Estrategias de optimización |
| Secuencias de intrones y exones de dos terminales. | Autoempalme de intrones catalizados por GTP para la ciclación de secuencias fuera del intrón. | Diseñado según el gen T4 td o gen precursor del ARNt del aceite de pescado. |
| Codificación | ires | Sitio de reconocimiento de ribosomas interno que regula la traducción del circRNA. | Detección de secuencias del sitio de entrada de ribosomas internos (IRES) de diferentes fuentes virales, por ejemplo, fuentes de EMCV y CVB3. |
| CDS | Regiones codificantes de proteínas, secuencias que codifican antígenos, anticuerpos u otras proteínas funcionales. | La optimización de codones aumenta el nivel de traducción; Ciertos codones no óptimos pueden desempeñar un papel en el plegamiento de proteínas. |
| sin codificar | Secuencias no codificantes | Apunta a miARN o proteínas para ejercer la regulación de genes o proteínas. | Dirigirse a sitios de unión específicos para miARN o proteínas puede repetir las secuencias del sitio de unión. |
Nuestras Características
- Combinación optimizada del sistema de ciclación PIE con estrategias de optimización razonables para lograr una tasa de ciclación de más del 80%;
- Equipo de optimización de CDS de vanguardia Cooperación con un equipo de algoritmos de IA profesional para completar la optimización de los codones de la región CDS;
- El circRNA de proceso maduro y perfecto se puede lograr con alta eficiencia de ciclación, alta estabilidad y alta eficiencia de traducción.
Estudio de caso
Yaohai Bio-Pharma lanzó el producto de control circRNA de proteína fluorescente verde mejorada (eGFP), que se basa en el sistema PIE para lograr la ciclación del ARN.
Utilizando un reactivo de transfección convencional, el ARNcirc de eGFP se transfecta en células 293T y la señal fluorescente de eGFP (verde) se puede detectar después de 24 h, que se mejorará después de 48 h. La señal fluorescente aún se puede detectar el día 7 y el día 14 después transfección.
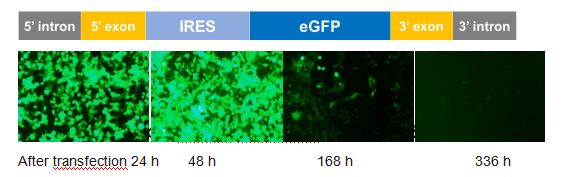
Validación de la expresión in vitro de circRNA eGFP

 EN
EN
 AR
AR
 HR
HR
 CS
CS
 DA
DA
 NL
NL
 FI
FI
 FR
FR
 DE
DE
 EL
EL
 IT
IT
 JA
JA
 KO
KO
 NO
NO
 PL
PL
 PT
PT
 RO
RO
 RU
RU
 ES
ES
 SV
SV
 IW
IW
 ID
ID
 LV
LV
 LT
LT
 SR
SR
 SK
SK
 SL
SL
 UK
UK
 VI
VI
 ET
ET
 HU
HU
 TH
TH
 TR
TR
 FA
FA
 AF
AF
 MS
MS
 BE
BE
 MK
MK
 UR
UR
 BN
BN