Caracterização de mRNA 3' poli(A)
Caracterização de mRNA 3' poli(A)
A cauda 3' poli(A) (poliadenosinas) do mRNA é essencial para a regulação pós-transcricional, influenciando a exportação, estabilidade e tradução do mRNA. No caso de mRNAs transcritos in vitro (IVT), a cauda poli(A) é introduzida durante a co-transcrição de maneira modelada ou através de um processo de cauda enzimática de maneira não modelada. Este 3 'poli (A) serve como um atributo crítico de qualidade (CQA) para RNAs IVT, necessitando de caracterização completa durante o desenvolvimento do processo e liberação do lote.
A Yaohai Bio-Pharma fornece serviços especializados de caracterização de 3' poli(A), empregando técnicas como clivagem de ribonuclease T1 (RNase T1), cromatografia líquida de alto desempenho de fase reversa de par de íons (IP-RP-HPLC)/cromatografia líquida-espectrometria de massa (LC-MS) e análise abrangente de dados.
Requisitos regulatórios para caracterização de mRNA 3' poli(A)
De acordo com as considerações regulamentares da OMS, “a integridade da estrutura do mRNA é considerada um atributo de qualidade crítico para a libertação do mRNA. Assim, o controle é necessário para a integridade do mRNA, eficiência de capeamento 5′, presença ou comprimento da cauda 3′ poli(A) e assim por diante.”
Método Analítico
Análise |
De Depósito |
Caracterização de mRNA 3' poli(A) |
LC-MS após digestão |
Procedimento
Passo 1. Clivagem de RNase T1
A análise direta do mRNA intacto completo por LC-MS é impraticável devido ao seu tamanho e heterogeneidade. Para examinar a cauda poli(A), ela deve ser clivada do mRNA completo. O método predominante envolve digestão enzimática com RNase T1, clivando seletivamente a extremidade 3 'de rG enquanto preserva a sequência poli (A). A amostra de mRNA sofre digestão com RNase T1, e fragmentos de clivagem contendo trechos poli(A) são isolados usando esferas magnéticas revestidas com oligo dT.
Etapa 2. IP-RP-HPLC/LC-MS
As caudas extraídas foram analisadas usando LC-MS, oferecendo informações precisas sobre o comprimento da cauda na resolução de um único nucleotídeo. Na análise de oligonucleotídeos LC-MS, separações de fase reversa de pareamento iônico (IP-RP) com aditivos de fase móvel de amina são o modo cromatográfico preferido, devido ao seu robusto poder de resolução e compatibilidade com espectrometria de massa (MS).
Passo 3. Análise de dados
O comprimento da cauda poli(A) e sua abundância relativa dependem do número e da intensidade dos picos divididos por uma massa de adenosina. Após o processamento ou desconvolução do espectro de massa, a massa antecipada de uma cauda de poli(A) de 100-meros (33,163 Da na caixa) é discernida, acompanhada por uma série de massas espaçadas por 329 amu, equivalente à massa de adenosina (A) .
Caso de caracterização 3' poli (A) com LC – MS
Yaohai-BioPharma desenvolveu um método robusto para caracterização de mRNA 3' poli(A), incluindo clivagem de RNase T1 e IP-RP-HPLC/LC-MS.
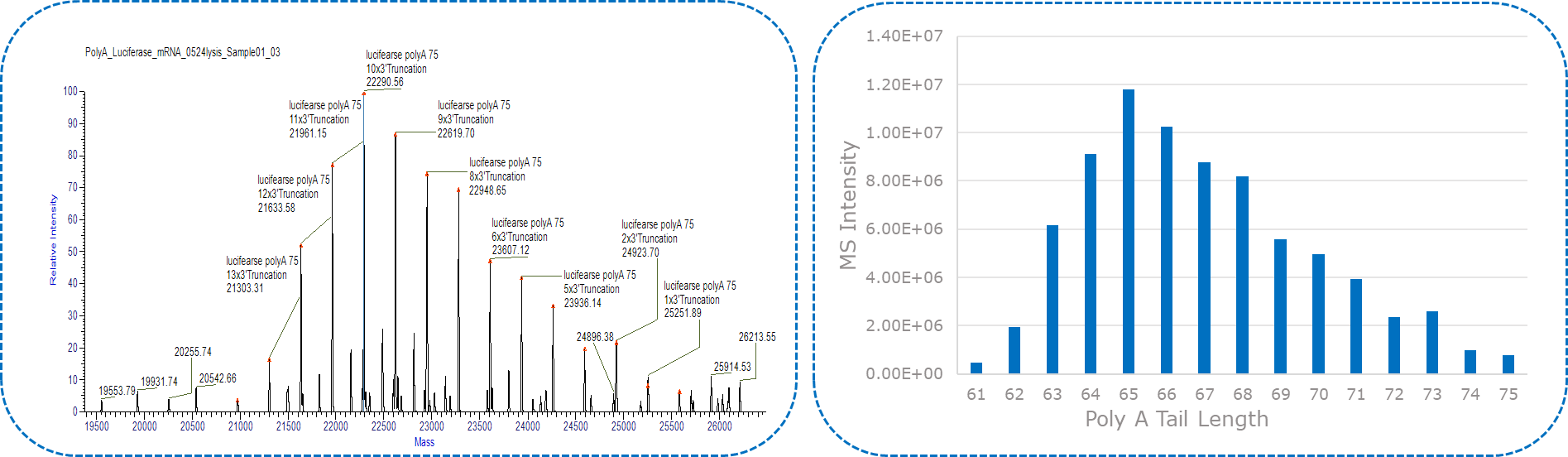
Yaohai-BioPharma desenvolveu um método robusto para caracterização de mRNA 3' poli(A), incluindo clivagem de RNase T1 e IP-RP-HPLC/LC-MS.

 EN
EN



