Caratterizzazione dell'mRNA 3' poli(A).
Caratterizzazione dell'mRNA 3' poli(A).
La coda 3' poli(A) (poliadenosine) dell'mRNA è parte integrante della regolazione post-trascrizionale, influenzando l'esportazione, la stabilità e la traduzione dell'mRNA. Nel caso degli mRNA trascritti in vitro (IVT), la coda poli(A) viene introdotta durante la co-trascrizione in modo basato su modelli o attraverso un processo di coda enzimatica in modo non basato su modelli. Questo poli(A) da 3′ funge da attributo critico di qualità (CQA) per gli RNA IVT, richiedendo una caratterizzazione approfondita sia durante lo sviluppo del processo che durante il rilascio del lotto.
Yaohai Bio-Pharma fornisce servizi specializzati di caratterizzazione del poli(A) 3', impiegando tecniche come la scissione della ribonucleasi T1 (RNasi T1), la cromatografia liquida ad alte prestazioni a fase inversa con coppia ionica (IP-RP-HPLC)/cromatografia liquida-spettrometria di massa (LC-MS) e analisi completa dei dati.
Requisiti normativi per la caratterizzazione dell'mRNA 3' poli(A).
Secondo le considerazioni normative dell'OMS, “l'integrità della struttura dell'mRNA è considerata un attributo di qualità critico per il rilascio dell'mRNA. Pertanto, è necessario il controllo dell’integrità dell’mRNA, dell’efficienza del capping 5′, della presenza o della lunghezza della coda poli(A) 3′ e così via.
Metodo analitico
Analisi |
Metodi |
Caratterizzazione dell'mRNA 3' poli(A). |
LC-MS dopo la digestione |
Procedura
Passaggio 1. Scissione della RNasi T1
L'analisi diretta dell'mRNA intatto a lunghezza intera mediante LC-MS non è pratica a causa delle sue dimensioni ed eterogeneità. Per esaminare la coda poli(A), è necessario separarla dall'mRNA a lunghezza intera. Il metodo prevalente prevede la digestione enzimatica con RNasi T1, che taglia selettivamente l'estremità 3' di rG preservando la sequenza poli(A). Il campione di mRNA viene sottoposto a digestione con RNasi T1 e i frammenti di scissione contenenti tratti di poli(A) vengono isolati utilizzando sfere magnetiche rivestite con oligo dT.
Passaggio 2. IP-RP-HPLC/LC-MS
Le code estratte sono state analizzate utilizzando LC-MS, offrendo informazioni precise sulla lunghezza della coda con risoluzione a singolo nucleotide. Nell'analisi LC-MS degli oligonucleotidi, le separazioni in fase inversa con accoppiamento ionico (IP-RP) con additivi della fase mobile amminica rappresentano la modalità cromatografica preferita, grazie al loro robusto potere risolvente e alla compatibilità con la spettrometria di massa (MS).
Passaggio 3. Analisi dei dati
La lunghezza della coda del poli(A) e la sua abbondanza relativa dipendono dal numero e dall'intensità dei picchi divisi per una massa di adenosina. Dopo l'elaborazione o la deconvoluzione dello spettro di massa, si distingue la massa prevista di una coda di poli(A) da 100 meri (33,163 Da nella scatola), accompagnata da una serie di masse distanziate di 329 amu, equivalenti alla massa di adenosina (A) .
Caso di caratterizzazione di poli (A) 3' con LC-MS
Yaohai-BioPharma ha sviluppato un metodo affidabile per la caratterizzazione del poli(A) dell'mRNA 3', compreso il clivaggio dell'RNasi T1 e l'IP-RP-HPLC/LC-MS.
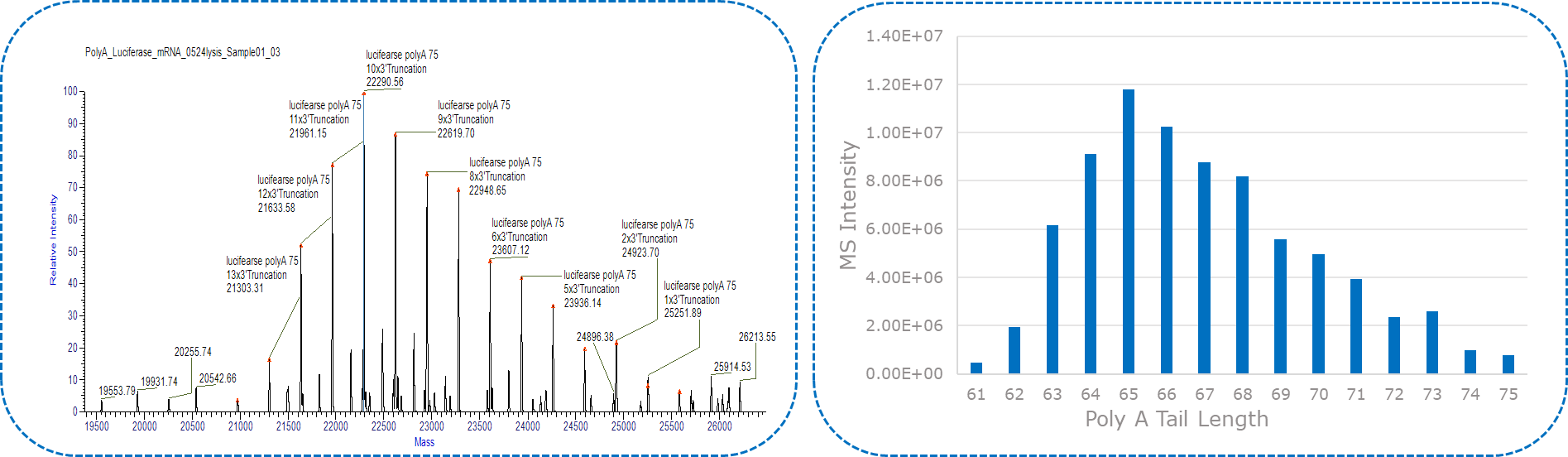
Yaohai-BioPharma ha sviluppato un metodo affidabile per la caratterizzazione del poli(A) dell'mRNA 3', compreso il clivaggio dell'RNasi T1 e l'IP-RP-HPLC/LC-MS.

 EN
EN



