Caracterización del ARNm 3' poli(A)
Caracterización del ARNm 3' poli(A)
La cola 3' poli(A) (poliadenosinas) del ARNm es parte integral de la regulación postranscripcional, lo que influye en la exportación, la estabilidad y la traducción del ARNm. En el caso de los ARNm transcritos in vitro (IVT), la cola poli(A) se introduce durante la cotranscripción mediante plantilla o mediante un proceso de cola enzimática sin plantilla. Este 3′ poli(A) sirve como atributo de calidad crítico (CQA) para los ARN IVT, lo que requiere una caracterización exhaustiva tanto durante el desarrollo del proceso como durante la liberación del lote.
Yaohai Bio-Pharma proporciona servicios especializados de caracterización de 3' poli(A), empleando técnicas como la escisión de ribonucleasa T1 (RNasa T1), cromatografía líquida de alto rendimiento de fase reversa de pares iónicos (IP-RP-HPLC)/cromatografía líquida-espectrometría de masas. (LC-MS) y análisis completo de datos.
Requisitos reglamentarios para la caracterización del ARNm 3' poli(A)
Según las consideraciones regulatorias de la OMS, “la integridad de la estructura del ARNm se considera un atributo de calidad crítico para la liberación del ARNm. Por lo tanto, se necesita control de la integridad del ARNm, la eficacia de la protección en 5', la presencia o longitud de la cola poli(A) en 3', etc.
Método analítico
Analisis | Métodos |
Caracterización del ARNm 3' poli(A) | LC-MS después de la digestión |
Procedimiento
Paso 1. Escisión de la RNasa T1
El análisis directo de ARNm intacto de longitud completa mediante LC-MS no es práctico debido a su tamaño y heterogeneidad. Para examinar la cola poli(A), se debe escindir del ARNm de longitud completa. El método predominante implica la digestión enzimática con RNasa T1, escindiendo selectivamente el extremo 3' de rG preservando al mismo tiempo la secuencia poli(A). La muestra de ARNm se somete a digestión con ARNasa T1 y los fragmentos de escisión que contienen tramos poli(A) se aíslan utilizando perlas magnéticas recubiertas de oligo dT.
Paso 2. IP-RP-HPLC/LC-MS
Las colas extraídas se sometieron a análisis mediante LC-MS, lo que ofrece información precisa sobre la longitud de la cola con una resolución de un solo nucleótido. En el análisis LC-MS de oligonucleótidos, las separaciones de fase reversa por emparejamiento iónico (IP-RP) con aditivos de fase móvil de amina son el modo cromatográfico preferido, debido a su sólido poder de resolución y compatibilidad con la espectrometría de masas (MS).
Paso 3. Análisis de datos
La longitud de la cola de poli(A) y su abundancia relativa dependen del número y la intensidad de los picos divididos por una masa de adenosina. Al procesar o desconvolucionar el espectro de masas, se discierne la masa anticipada de una cola poli(A) de 100 meros (33,163 Da en caja), acompañada de una serie de masas espaciadas por 329 uma, equivalente a la masa de adenosina (A). .
Caso de caracterización de poli(A) 3' con LC-MS
Yaohai-BioPharma ha desarrollado un método sólido para la caracterización de poli(A) 3' de ARNm, incluida la escisión de RNasa T1 e IP-RP-HPLC/LC-MS.
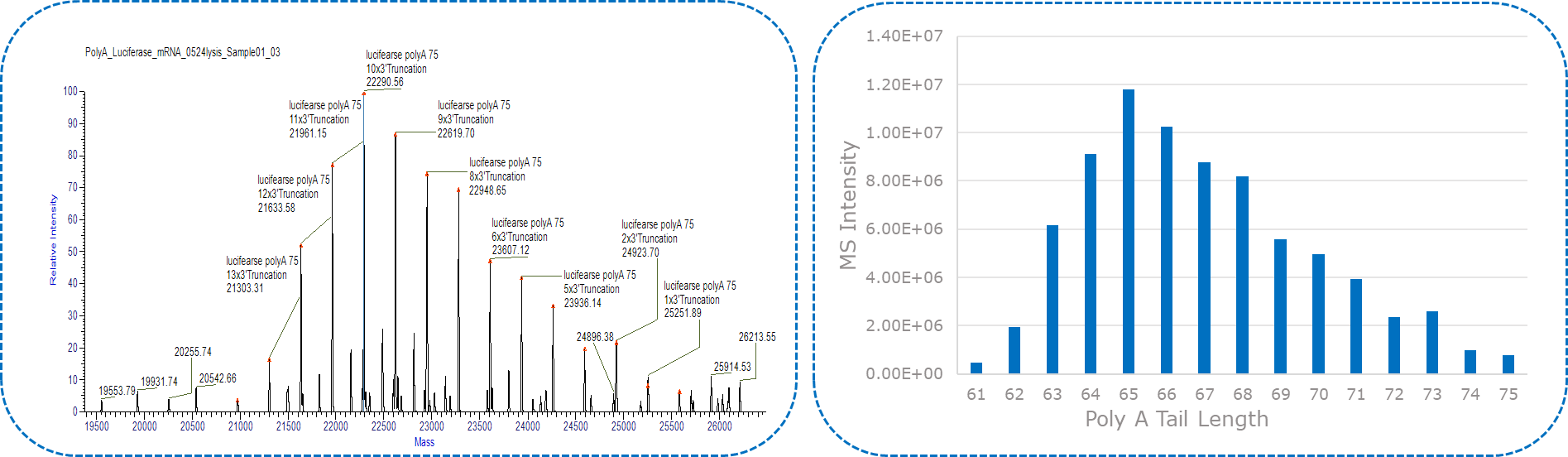
Yaohai-BioPharma ha desarrollado un método sólido para la caracterización de poli(A) 3' de ARNm, incluida la escisión de RNasa T1 e IP-RP-HPLC/LC-MS.

 EN
EN



