A Yaohai Bio-Pharma prepara circRNA com base no sistema PIE (alinhamento de exons e introns), que se apoia na função autosplicante dos introns do tipo I para alcançar a ciclização do RNA. A estrutura PIE é projetada usando o gene T4 td ou o gene precursor de tRNA peixe, e a disposição é a seguinte:
O intron de RNA e o fragmento de exon de suporte são divididos em duas partes (extremidade 5' e extremidade 3'), onde a sequência da extremidade 5' é transferida para a cauda da sequência alvo, a sequência da extremidade 3' é inserida na frente da sequência alvo, e a sequência do gene alvo é inserida no meio.
Sob a catalisação do GTP, a estrutura PIE leva à ciclização de sequências que não sejam introns. Combinado com uma estratégia razoável de aumento da taxa de ciclização, a Yaohai Bio-Pharma pode alcançar a ciclização de sequências de até 4 kb com uma taxa de ciclização superior a 80%.
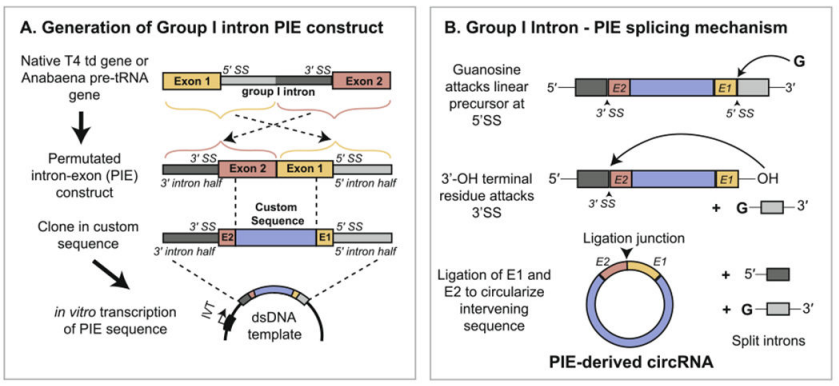
Figura 1. Circulação in vitro de circRNA com base no sistema PIE
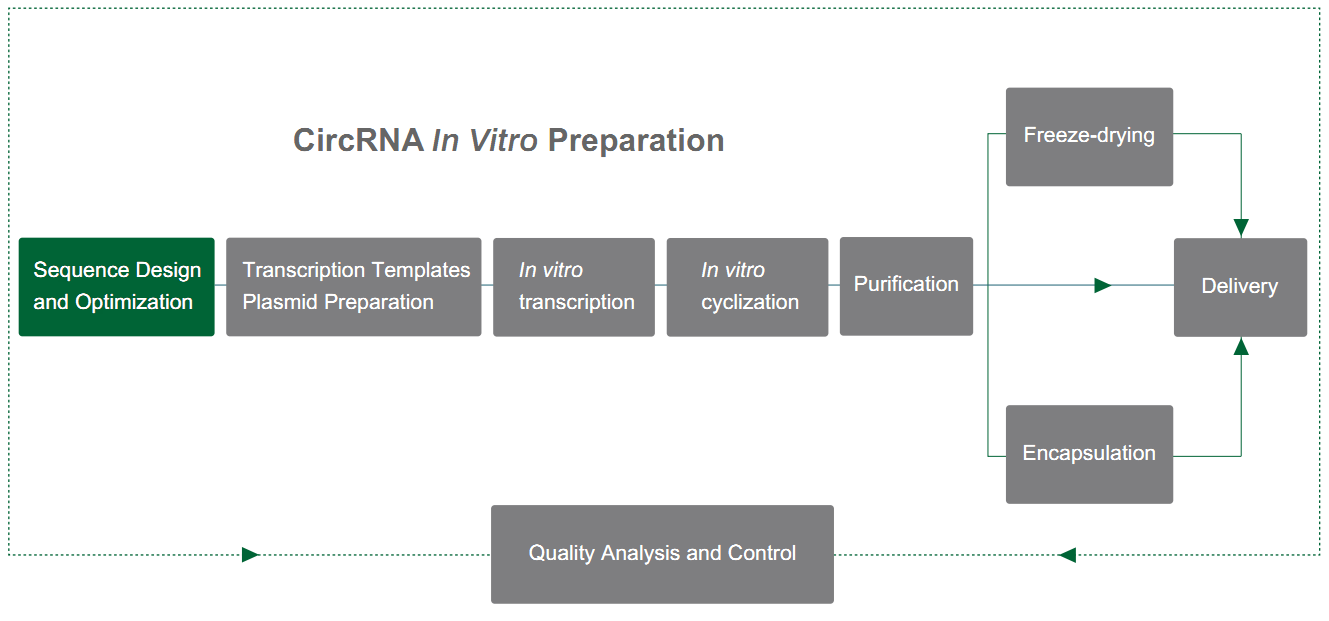
Detalhes dos Serviços
| Processo |
Serviço Opcional |
Detalhes do Serviço |
Prazo de Entrega (dia útil) |
| design e otimização de sequências de circRNA |
Design e otimização de sequências codificantes |
Alinhamento de sequências CDS
Otimização de codons CDS
|
1 |
| Design e otimização de sequências não codificantes |
Design e otimização de sequências de introns e exons
Design e otimização de sequências de braços homólogos
Design e otimização de sequências espaçadoras
|
1-2 |
Estratégias Comuns para o Design de Sequências de mRNA
| Componentes de CircRNA |
Funções Biológicas |
Estratégias de Otimização |
| Sequências de introns e exons terminais |
Auto-splicagem catalisada por GTP para ciclização de sequências fora do intron. |
Projetado com base no gene T4 td ou no gene precursor de tRNA de óleo de peixe. |
| Codificação |
IRES |
Sítio de reconhecimento interno do ribossomo que regula a tradução de circRNA. |
Triagem de sequências de sítios de entrada internos do ribossomo (IRES) de diferentes fontes virais, por exemplo, fontes EMCV, CVB3. |
| CDS |
Regiões codificadoras de proteínas, sequências codificadoras de antígenos, anticorpos ou outras proteínas funcionais. |
A otimização de codons aumenta o nível de tradução; certos codons não ótimos podem desempenhar um papel na dobra de proteínas. |
| Não codificante |
Sequências não codificantes |
Alvejar miRNAs ou proteínas para exercer regulação gênica ou de proteínas. |
Alvejar sítios de ligação específicos para miRNAs ou proteínas pode repetir as sequências do sítio de ligação. |
Nossos Recursos
- Sistema Otimizado de Ciclização PIE combinado com estratégias de otimização razoáveis para alcançar uma taxa de ciclização de mais de 80%;
- Colaboração da Equipe de Otimização de CDS de Ponta com uma equipe profissional de algoritmos de IA para completar a otimização dos codons da região CDS;
- Processo Maduro e Perfeito circRNA pode ser alcançado com alta eficiência de ciclização, alta estabilidade e alta eficiência de tradução.
Estudo de Caso
Yaohai Bio-Pharma lançou o produto de controle proteína de fluorescência verde aprimorada (eGFP) circRNA, que é baseado no sistema PIE para alcançar a ciclização do RNA.
Usando um reagente de transfeção convencional, o circRNA eGFP é transfetado em células 293T e o sinal fluorescente de eGFP (verde) pode ser detectado após 24h, sendo intensificado após 48h. O sinal fluorescente ainda pode ser detectado no 7º e no 14º dia após a transfeção.
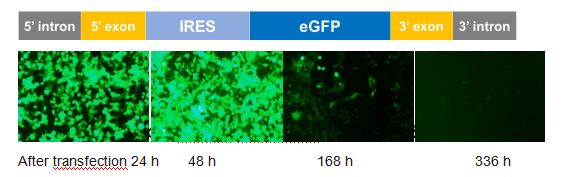
Validação da expressão in vitro do circRNA eGFP

 EN
EN
 AR
AR
 HR
HR
 CS
CS
 DA
DA
 NL
NL
 FI
FI
 FR
FR
 DE
DE
 EL
EL
 IT
IT
 JA
JA
 KO
KO
 NO
NO
 PL
PL
 PT
PT
 RO
RO
 RU
RU
 ES
ES
 SV
SV
 IW
IW
 ID
ID
 LV
LV
 LT
LT
 SR
SR
 SK
SK
 SL
SL
 UK
UK
 VI
VI
 ET
ET
 HU
HU
 TH
TH
 TR
TR
 FA
FA
 AF
AF
 MS
MS
 BE
BE
 MK
MK
 UR
UR
 BN
BN