Yaohai Bio-Pharma는 RNA 고리화를 달성하기 위해 유형 I 인트론의 자가 접합 기능에 의존하는 PIE 시스템(엑손과 인트론의 정렬)을 기반으로 circRNA를 준비합니다. PIE 구조는 T4 td 유전자 또는 fishy tRNA 전구체 유전자를 이용하여 설계되었으며 그 배열은 다음과 같다.
RNA 인트론과 지지 엑손 단편은 두 부분(5' 말단과 3' 말단)으로 나뉘는데, 여기서 5' 말단 서열은 표적 서열의 꼬리 부분으로 전달되고, 3' 말단 서열은 표적 서열의 앞쪽에 삽입된다. 표적 서열, 표적 유전자 서열이 중간에 삽입된다.
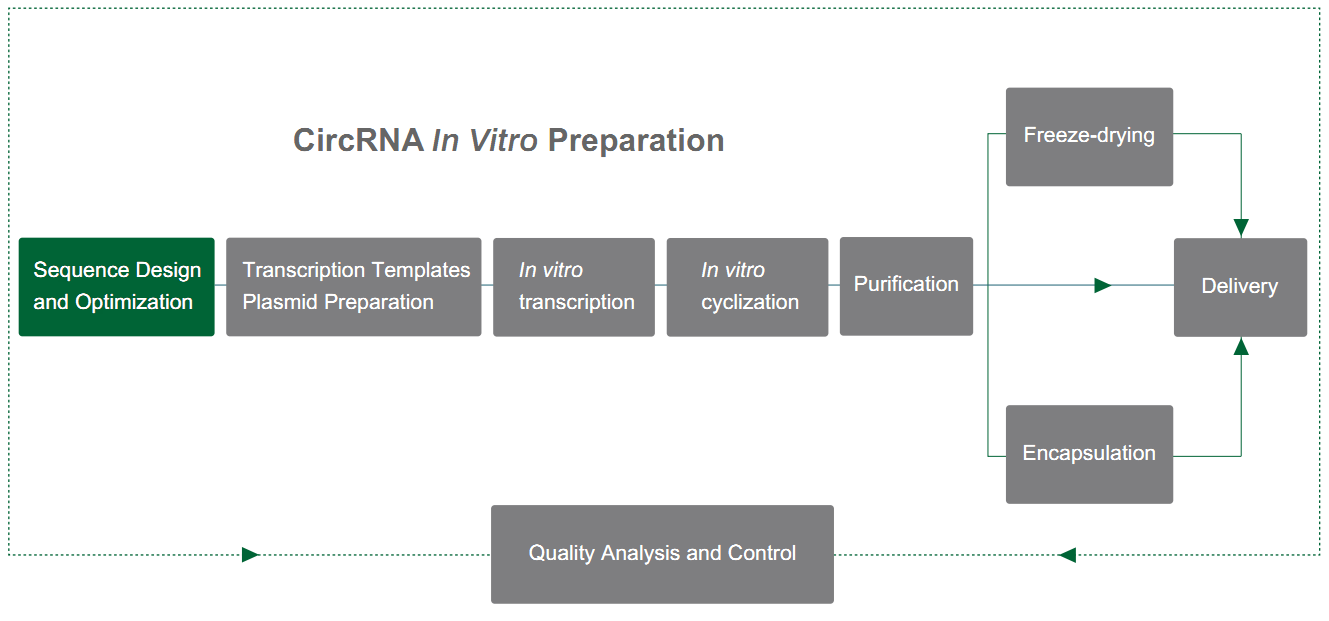
GTP의 촉매작용 하에서 PIE 구조는 인트론 이외의 서열의 고리화를 유도합니다. Yaohai Bio-Pharma는 합리적인 고리화 속도 향상 전략과 결합하여 4% 이상의 고리화 속도로 최대 80kb까지 서열의 고리화를 달성할 수 있습니다.
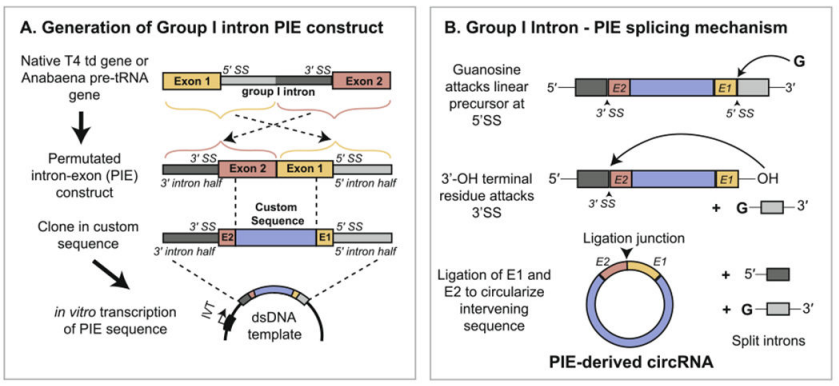
그림 1. PIE 시스템을 기반으로 한 circRNA의 시험관 내 순환
| 방법 | 선택서비스 | 서비스 내용 | 배송기간(평일) |
| circRNA 서열 설계 및 최적화 | 코딩 시퀀스의 설계 및 최적화 |
CDS 서열 정렬 CDS 코돈 최적화 |
1 |
| 비코딩 시퀀스의 설계 및 최적화 |
인트론 및 엑손 서열 설계 및 최적화 상동 암 시퀀스 설계 및 최적화 스페이서 시퀀스 설계 및 최적화 |
1-2 |
| CircRNA 구성 요소 | 생물학적 기능 | 최적화 전략 | |
| 2말단 인트론 및 엑손 서열 | 인트론 외부 서열의 고리화를 위한 GTP-촉매된 인트론 자가-접합. | T4 td 유전자 또는 어유 tRNA 전구체 유전자에 따라 설계되었습니다. | |
| 코딩 | IRES | circRNA의 번역을 조절하는 내부 리보솜 인식 부위. | 다양한 바이러스 소스(예: EMCV, CVB3 소스)로부터 내부 리보솜 진입 부위(IRES) 서열을 스크리닝합니다. |
| CDS | 단백질 코딩 영역, 항원, 항체 또는 기타 기능성 단백질을 코딩하는 서열. | 코돈 최적화는 번역 수준을 높입니다. 특정 비최적 코돈은 단백질 접힘에 역할을 할 수 있습니다. | |
| 비코딩 | 비코딩 서열 | 유전자 또는 단백질 조절을 발휘하기 위해 miRNA 또는 단백질을 표적으로 삼습니다. | miRNA 또는 단백질에 대한 특정 결합 부위를 표적으로 삼으면 결합 부위의 서열이 반복될 수 있습니다. |
Yaohai Bio-Pharma는 RNA의 고리화를 달성하기 위해 PIE 시스템을 기반으로 하는 대조 제품인 향상된 녹색 형광 단백질(eGFP) circRNA를 출시했습니다.
기존의 형질감염 시약을 사용하여 eGFP circRNA가 293T 세포에 형질감염되고 eGFP(녹색) 형광 신호는 24시간 후에 감지될 수 있으며 이는 48시간 후에 향상됩니다. 형광 신호는 7일째와 14일 후에도 여전히 감지될 수 있습니다. 형질감염.
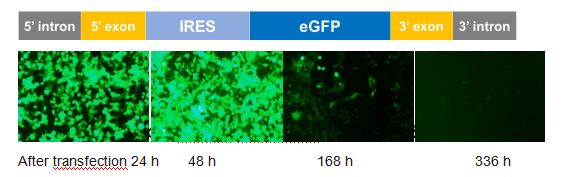
eGFP circRNA의 시험관 내 발현 검증