Caractérisation de l'ARNm 3' poly(A)
Caractérisation de l'ARNm 3' poly(A)
La queue 3' poly(A) (polyadénosines) de l'ARNm fait partie intégrante de la régulation post-transcriptionnelle, influençant l'exportation, la stabilité et la traduction de l'ARNm. Dans le cas des ARNm transcrits in vitro (IVT), la queue poly(A) est introduite soit lors de la co-transcription selon un modèle, soit via un processus de queue enzymatique sans modèle. Ce 3' poly(A) sert d'attribut de qualité critique (CQA) pour les ARN IVT, nécessitant une caractérisation approfondie pendant le développement du processus et la libération des lots.
Yaohai Bio-Pharma fournit des services spécialisés de caractérisation du 3' poly(A), employant des techniques telles que le clivage de la ribonucléase T1 (RNase T1), la chromatographie liquide haute performance en phase inversée par paire d'ions (IP-RP-HPLC)/chromatographie liquide-spectrométrie de masse. (LC-MS) et analyse complète des données.
Exigences réglementaires pour la caractérisation de l'ARNm 3' poly(A)
Selon les considérations réglementaires de l’OMS, « l’intégrité de la structure de l’ARNm est considérée comme un attribut de qualité critique pour la libération de l’ARNm. Ainsi, un contrôle est nécessaire pour l’intégrité de l’ARNm, l’efficacité du coiffage 5’, la présence ou la longueur de la queue poly(A) 3’, etc.
Méthode analytique
Analyse |
Méthodologie |
Caractérisation de l'ARNm 3' poly(A) |
LC-MS après digestion |
Procédure
Étape 1. Clivage de la RNase T1
L’analyse directe de l’ARNm intact de pleine longueur par LC-MS n’est pas pratique en raison de sa taille et de son hétérogénéité. Pour examiner la queue poly(A), elle doit être séparée de l’ARNm complet. La méthode répandue implique une digestion enzymatique avec la RNase T1, clivant sélectivement l'extrémité 3' de rG tout en préservant la séquence poly(A). L'échantillon d'ARNm subit une digestion avec la RNase T1 et les fragments de clivage contenant des tronçons poly(A) sont isolés à l'aide de billes magnétiques recouvertes d'oligo dT.
Étape 2. IP-RP-HPLC/LC-MS
Les queues extraites ont été analysées par LC-MS, offrant des informations précises sur la longueur des queues avec une résolution mononucléotidique. Dans l'analyse LC-MS des oligonucléotides, les séparations en phase inversée par appariement d'ions (IP-RP) avec des additifs de phase mobile d'amine constituent le mode chromatographique préféré, en raison de leur pouvoir de résolution robuste et de leur compatibilité avec la spectrométrie de masse (MS).
Étape 3. Analyse des données
La longueur de la queue poly(A) et son abondance relative dépendent du nombre et de l'intensité des pics divisés par une masse d'adénosine. Lors du traitement ou de la déconvolution du spectre de masse, la masse anticipée d'une queue poly(A) 100-mer (33,163 329 Da dans la boîte) est discernée, accompagnée d'une série de masses espacées de XNUMX amu, équivalentes à la masse de l'adénosine (A) .
Cas de caractérisation 3' poly(A) avec LC-MS
Yaohai-BioPharma a développé une méthode robuste pour la caractérisation de l'ARNm 3' poly(A), y compris le clivage de la RNase T1 et l'IP-RP-HPLC/LC-MS.
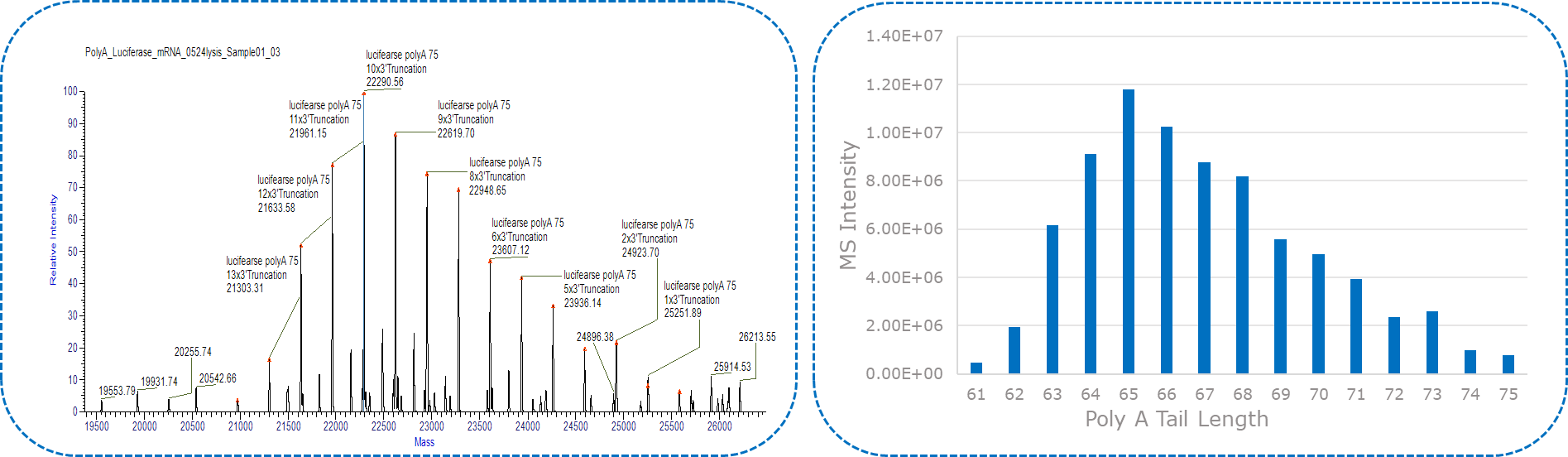
Yaohai-BioPharma a développé une méthode robuste pour la caractérisation de l'ARNm 3' poly(A), y compris le clivage de la RNase T1 et l'IP-RP-HPLC/LC-MS.

 EN
EN



