Yaohai Bio-Pharma bereidt circRNA voor op basis van het PIE-systeem (uitlijning van exonen en intronen), wat leunt op de zelf-splicing functie van type I introns om RNA-cyclisatie te realiseren. De PIE-structuur wordt ontworpen met behulp van het T4 td-gen of het visachtige tRNA-precursor-gen, en de indeling is als volgt:
Het RNA-intron en het ondersteunende exongedeelte worden verdeeld in twee delen (5' terminaal en 3' terminaal), waarbij de 5' terminale sequentie wordt overgedragen naar het einde van de doelsequentie, de 3' terminale sequentie wordt ingevoegd voor de doelsequentie, en de doelgeensequentie wordt in het midden geïnsert.
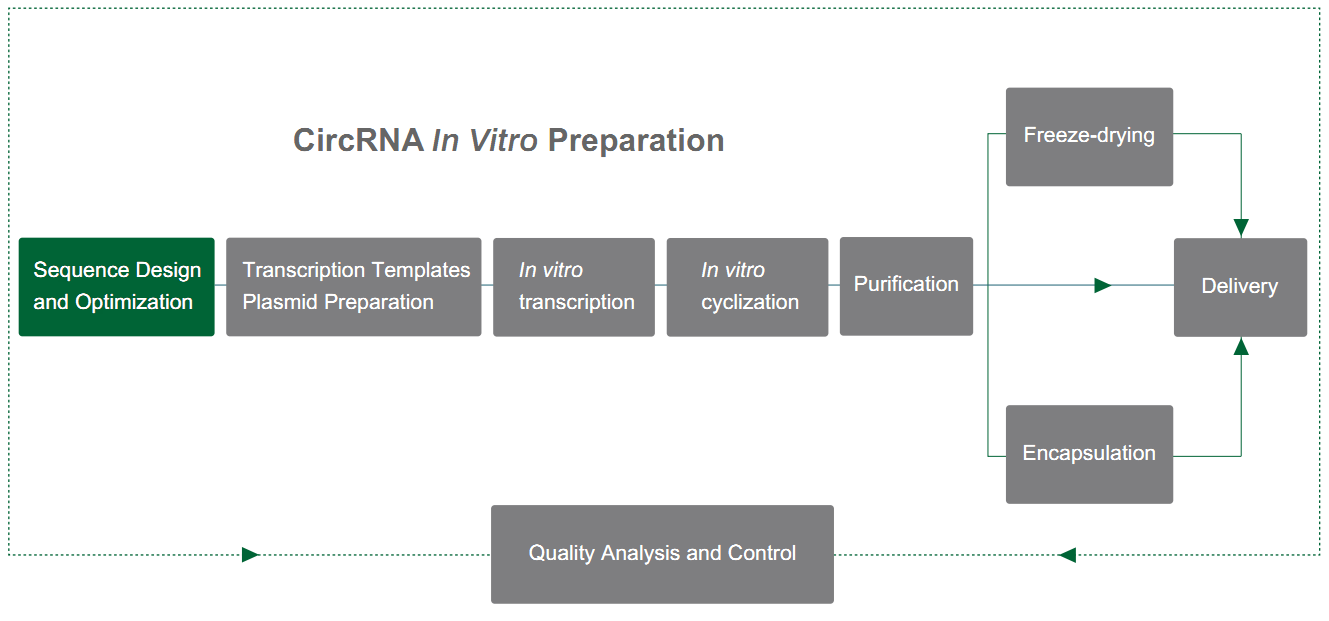
Onder de katalyse van GTP leidt de PIE-structuur tot de cyclisatie van sequenties die niet corresponderen met introns. In combinatie met een redelijke strategie voor het verbeteren van de cyclisatiesnelheid kan Yaohai Bio-Pharma cyclisatie bereiken van sequenties tot 4 kb met een cyclisatiefrequentie van meer dan 80%.
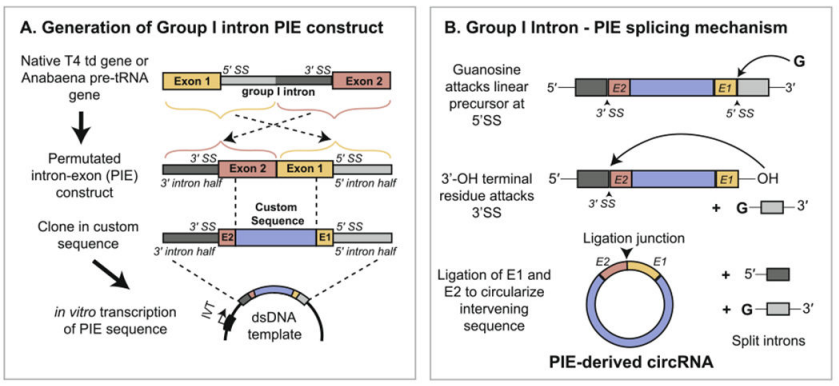
Figuur 1. In vitro cyclisatie van circRNA gebaseerd op het PIE-systeem
Service Details
| Proces |
Optionele Diensten |
Service Details |
Leveringsperiode (werkdag) |
| circRNA sequentiëringontwerp en optimalisatie |
Ontwerp en optimalisatie van coderende sequenties |
CDS sequentieuitlijning
CDS Codon Optimalisatie
|
1 |
| Ontwerp en optimalisatie van niet-coderende volgordes |
Intron en exon sequentieontwerp en optimalisatie
Ontwerp en optimalisatie van homologe arm sequenties
Ontwerp en optimalisatie van spacer sequenties
|
1-2 |
Gemeenschappelijke strategieën voor mRNA-sequentieontwerp
| CircRNA-componenten |
Biologische functies |
Optimalisatiestrategieën |
| Tweeterminal intron- en exonsequenties |
GTP-gecatalyseerde intron zelf-splicing voor cyclisatie van sequenties buiten het intron. |
Ontworpen volgens de T4 td-gene of de visolie tRNA-precursor-gene. |
| Codering |
IRES |
Interne ribosoomherkenningssite die de vertaling van circRNA reguleert. |
Screening van interne ribosoomentrysites (IRES)-sequenties uit verschillende virale bronnen, bijv. EMCV, CVB3 bronnen. |
| CDS |
Proteïne-coderende gebieden, sequenties die coderen voor antigenen, antilichamen of andere functionele proteïnen. |
Codonoptimalisatie verhoogt het niveau van vertaling; bepaalde niet-optimale codons kunnen een rol spelen in de vouwing van proteïnen. |
| Niet-coderend |
Niet-coderende sequenties |
Doel miRNAs of proteïnen om gen- of proteïneregulatie uit te oefenen. |
Richten op specifieke bindingsplaatsen voor miRNAs of proteïnen kan de sequenties van de bindingsplaats herhalen. |
Onze functies
- Goptimaliseerd PIE Cyclisatiesysteemcombinatie met redelijke optimalisatiestrategieën om een cyclisatieratio van meer dan 80% te bereiken;
- Scherpe CDS Optimalisatie Team Samenwerking met een professioneel AI algoritme team om de optimalisatie van CDS regio codons af te ronden;
- Volwassen en Perfect Proces circRNA kan bereikt worden met een hoge cyclisatie efficiëntie, hoge stabiliteit en hoge vertaalefficiëntie.
Gevalstudie
Yaohai Bio-Pharma lanceerde het controleproduct versterkte groene fluorescerende eiwit (eGFP) circRNA, dat gebaseerd is op het PIE systeem om de cyclisatie van RNA te realiseren.
Met behulp van een conventioneel transfectiereagens wordt eGFP circRNA getransfereerd in 293T cellen, en na 24 uur kan er een eGFP (groen) fluorescerend signaal gedetecteerd worden, dat na 48 uur versterkt wordt. Het fluorescerende signaal kan nog steeds gedetecteerd worden op de 7e dag en de 14e dag na transfectie.
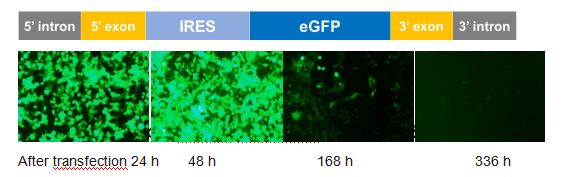
In vitro expressievalidatie van eGFP circRNA

 EN
EN
 AR
AR
 HR
HR
 CS
CS
 DA
DA
 NL
NL
 FI
FI
 FR
FR
 DE
DE
 EL
EL
 IT
IT
 JA
JA
 KO
KO
 NO
NO
 PL
PL
 PT
PT
 RO
RO
 RU
RU
 ES
ES
 SV
SV
 IW
IW
 ID
ID
 LV
LV
 LT
LT
 SR
SR
 SK
SK
 SL
SL
 UK
UK
 VI
VI
 ET
ET
 HU
HU
 TH
TH
 TR
TR
 FA
FA
 AF
AF
 MS
MS
 BE
BE
 MK
MK
 UR
UR
 BN
BN